正所谓前人栽树,后人乘凉。
感谢铁汉提供的有关隐马尔科夫模型的资料。
其他参考来源:《HMMER3.2 Userguide》。
第一部分 隐马尔科夫模型(HMM)
一、什么是熵
熵表示物质系统状态的一种度量,用它来表征系统的无序程度。熵越大,系统越无序。熵可以表示任何一种能量在空间中分布的均匀程度,能量分布得越均匀,熵就越大。熵表现了一个系统所处状态的不确定性程度。香农在描述一个信息系统的时候就借用了熵的概念,这里的“熵”表示的是这个信息系统的平均信息量(平均不确定程度)。
二、最大熵模型
最大熵原理:保留全部的不确定性,将风险降到最小。(不要把所有的鸡蛋放在篮子里,这样可以降低风险)
三、HMM(隐马尔科夫模型)
HMM是用来描述隐含未知参数的统计模型,其难点是从可观察的参数中确定该过程的隐含参数,然后利用这些参数来作进一步的分析,例如模式识别。
举一个经典的举一个经典的例子:
一个东京的朋友每天根据天气{下雨,天晴}决定当天的活动{公园散步,购物,清理房间}中的一种,我每天只能在twitter上看到她发的推“啊,我前天公园散步、昨天购物、今天清理房间了!”,那么我可以根据她发的推特推断东京这三天的天气。在这个例子里,显状态是活动,隐状态是天气。
四、HMM模型能解决的问题
使用HMM模型时我们的问题一般有这两个特征:
1)我们的问题是基于序列的,比如时间序列,或者状态序列。
2)我们的问题中有两类数据,一类序列数据是可以观测到的,即观测序列;而另一类数据是不能观察到的,即隐藏状态序列,简称状态序列。 有了这两个特征,那么这个问题一般可以用HMM模型来尝试解决。这样的问题在实际生活中是很多的。比如:我现在在打字写博客,我在键盘上敲出来的一系列字符就是观测序列,而我实际想写的一段话就是隐藏序列,输入法的任务就是从敲入的一系列字符尽可能的猜测我要写的一段话,并把最可能的词语放在最前面让我选择,这就可以看做一个HMM模型了。再举一个,我在和你说话,我发出的一串连续的声音就是观测序列,而我实际要表达的一段话就是状态序列,你大脑的任务,就是从这一串连续的声音中判断出我最可能要表达的话的内容。
从这些例子中,我们可以发现,HMM模型可以无处不在。
第二部分 Hmmer软件的使用
简单的讲HMMER就是一款比对软件,可以类似于blast等,但是它提供的比对结果要精确于blast,相应的速度也要慢。与其类似的比对软件有PSI-blast、SAM、PFTOOLS等。
用途一:对于一条未知的序列在蛋白质数据库中寻找比对,HMMER完全可以取代目前常用的BLASTP 和 PSI-BLAST。
用途二:可以自动注释蛋白质结构域。包括pfam、SMART。
用途三:在数据库中寻找已有家族的相似序列。
一、hmmer包含的程序
phmmer: 与Blastp类似,使用一个蛋白质序列搜索蛋白质序列库;对于只有一条未知蛋白序列的情况下,就省去多重序列比对和建立模型的过程直接一步到位,期间使用了BLOSUM62 scores打分矩阵,类似于BLASTP-like
jackhmmer: 与psiBlast类似,蛋白质序列迭代搜索蛋白质序列库;
hmmbuild: 用多重比对序列构建HMM模型(建立参考数据的隐马尔可科夫模型);
hmmsearch: 使用HMM模型搜索序列库(在数据库中寻找已经建立好的模体);
hmmscan: 使用序列搜索HMM库;
hmmalign: 使用HMM为线索,构建多重比对序列;
hmmconvert: 转换HMM格式
hmmemit: 从HMM模型中,得到一个模式序列;
hmmfetch: 通过名字或者接受号从HMM库中取回一个HMM模型;
hmmpress:格式化HMM数据库,以便于hmmscan搜索使用;
hmmstat: 显示HMM数据库的统计信息;
二、使用HMM模型搜索序列数据库
1.使用hmmbuild构建HMM模型
输入:多重序列比对的文件(Stockholm file)
输出:建立的这些多重序列比对的隐马尔可科夫模型,
例子: hmmbuild globins4.hmm tutorial/globins4.sto
globins4.hmm为输出的HMM模型
2.使用hmmsearch搜索蛋白质序列数据库
输入:建立好的参考数据模型、要搜索的数据库
输出:输出结果
例子: hmmsearch globins4.hmm uniprot sprot.fasta > globins4.out
globins4.out为输出的结果文件结果如下: 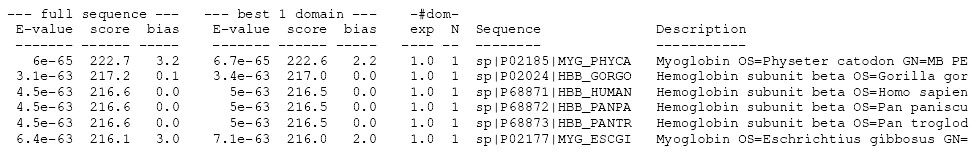
三、使用蛋白质序列搜索HMM数据库(参考我本人做的工作)
构建HMM数据库,HMM数据库是包含多个HMM模型的文件,可以从Pfam、SMART、TIGRFams下载,也可以自己由多重比对序列集中构建,如:
> hmmbuild NBS-LRR_seed.hmm sample.stockholm
使用hmmpress格式化数据库,包括压缩以及创建索引,命令如下:
> hmmpress minifam 这个步骤可以很快的执行完成,输出的内容如下:
Working… done.
Pressed and indexed 3 HMMs (3 names and 2 accessions).
Models pressed into binary file: minifam.h3m
SSI index for binary model file: minifam.h3i
Profiles (MSV part) pressed into: minifam.h3f
Profiles (remainder) pressed into: minifam.h3p 使用hmmscan搜索HMM数据库,命令如下:
> hmmscan minifam tutorial/7LESS_DROME 输出如下:
