正所谓前人栽树,后人乘凉。
感谢思考问题的熊博士提供的详细而全面的资料。
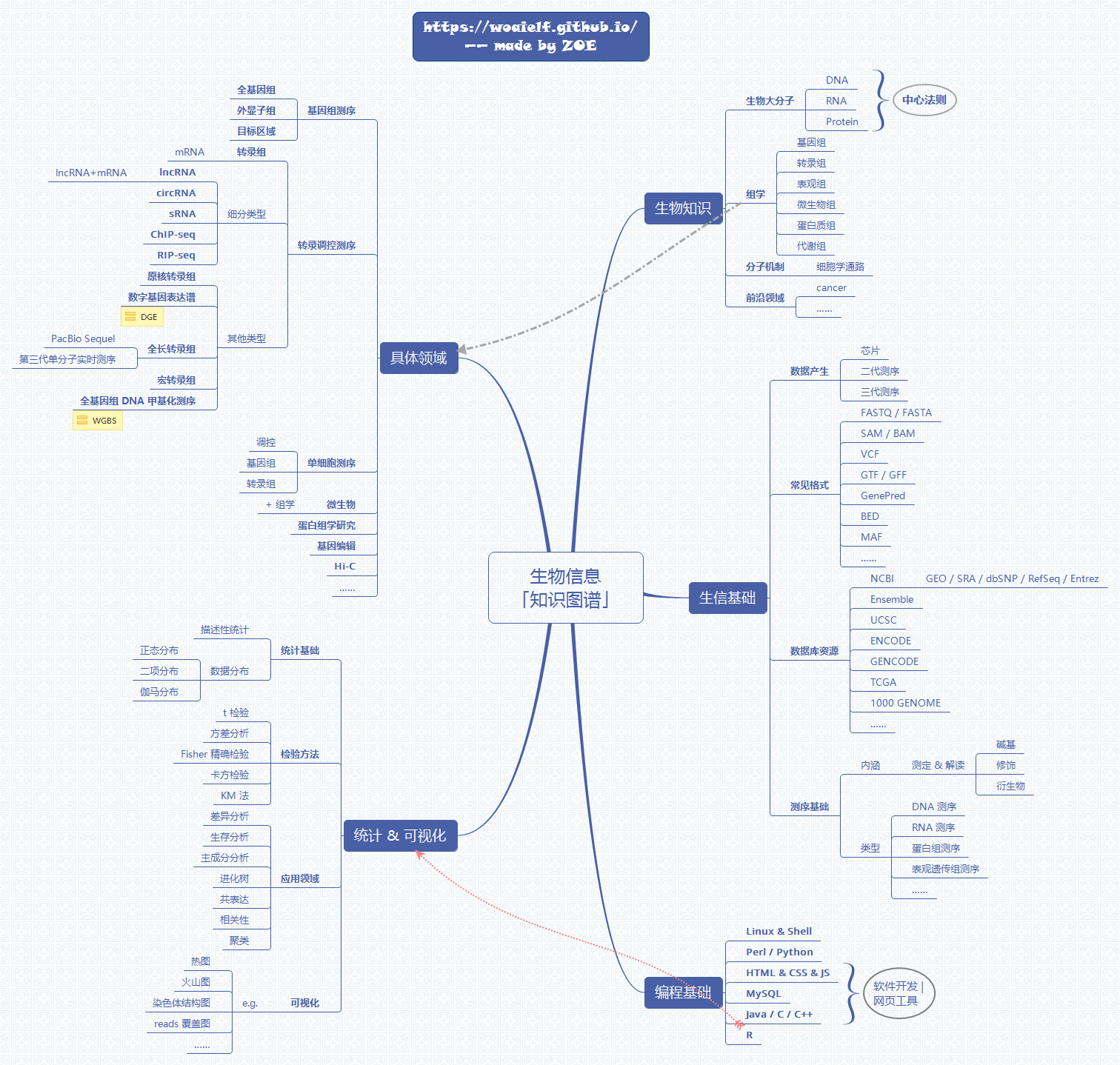
1. 前言
首先:通过可视化方法来熟练使用基于linux系统的服务器,
接着:实践两三个NGS组学数据分析流程,实践过程需要学习获得数据格式,数据库资源等生物信息学背景。
在此基础上:学习perl/python/R的编程基础,并应用于NGS组学数据分析的下游,个性化分析。
2. 服务器使用
基本配置是8G内存+0.5T硬盘(配置不达标问题也不大),系统可以是ubuntu,centos等linux系统,或者 MAC,或者改进版的Windows系统。
2.1 linux系统
推荐使用ubuntu系统,可视化做的比较好,对新手比较友好。
2.2 MAC系统
如果是苹果电脑,那么非常适合做生物信息学数据分析。需要安装brew,textwranger,filezilla
2.3 windows电脑
大多数人都是这个操作系统,如果是Windows10系统,那么开始其内置的ubuntu即可。如果是其它Windows版本,就需要安装虚拟机,在虚拟机里面安装ubuntu,centos等linux系统。
对于Windows用户还需要下载安装好 xshell,winscp,everything,notepad++,git或者同等功能的软件。
2.4 三系统共用软件
无论是哪种电脑操作系统,都需要安装anaconda,pycharm,R/Rstudio,typora
上面提到的软件的简介和部分网址如下:
- anaconda 为了学习Python,conda自带科学计算的python包(安装Python3.6.x版本的);
- pycharm community(三平台通用的 ),Python学习的IDE(社区版);
- git 虚拟shell界面,为了在window下使用一些shell命令,自带Perl;
- R/Rstudio 为了学习R语言来进行数据处理和统计绘图(安装最新版本);
- Xshell 远程连接服务器 (安装时点击免费版) ;
- Winscp 为了本地与远程服务器间安全的复制文件;
- Xming 为了图形化显示远程服务器的应用、显示图片、浏览器等;
- Vmware 为了在windows下面安装Linux虚拟机,同时选择一个国内镜像下载自己喜欢的Linux版本ios镜像;
- Everything 为了可以在windows下快速找到文件;
- notepad++ 为了可以在windows下编写脚本或查看文本文件;
- ActiveState/Strawberry Perl 为了在windows下学习Perl,但git自带Perl,可以选择不安装;
- 有道/为知云笔记/印象笔记 记录学习笔记,构建知识体系(建议使用两个云笔记软件,一个用于原创,一个用于网络资料收集);
-
typora 学习markdown语法,查看老师提供的md格式的讲义;
- perl https://www.activestate.com/activeperl/downloads
- miniconda https://conda.io/miniconda.html
- R https://cloud.r-project.org/
- rstudio https://www.rstudio.com/products/rstudio/download/
- Xshell https://www.netsarang.com/products/xsh_overview.html
-
winscp https://winscp.net/eng/docs/lang:chs
- ubuntu http://cn.ubuntu.com/download/
- 虚拟机管理工具 vmware workstation https://www.vmware.com/products/workstation.html
3. 去可视化熟悉linux命令
- 鸟哥的linux私房菜 (入门重点看4-12章即可)
- 在线查询linux命令
- http://man.linuxde.net/
- https://jaywcjlove.github.io/linux-command/
- 在线学习网站
- https://www.shiyanlou.com/courses/1
- http://www.imooc.com/learn/175
- http://www.runoob.com/linux/linux-tutorial.html
4. 生物信息入门基本知识
4.1 数据产生
- 产生方式
- arrary
- genome tiling arrary
- high throughput sequencing
- snp arrary
- protein arrary
- 数据种类
- expression profiling
- genome binding/occupancy profiling
- genome variation profiling
- methylation profiling
- non coding rna profiling
- protein profiling
- snp genotyping
- 二代测序
- 454
- SOLiD
- illumina https://www.illumina.com.cn/ 给出illumina官网的地址是因为里面资料很多。
- 三代测序
- PacBio RS
- Nanopore MinlON
4.2 数据存储类型
- 学习网址 https://genome.ucsc.edu/FAQ/FAQformat.html
- FASTA
- FASTQ
- GFF/GTF
- SAM
- BAM
- CRAM
- VCF
- 其他
4.3 数据库资源
- 拟南芥
- TAIR https://www.arabidopsis.org/
- araport https://www.araport.org/
- 水稻
- rapdb http://rapdb.dna.affrc.go.jp/
- msu http://rice.plantbiology.msu.edu/
- ricexpro http://ricexpro.dna.affrc.go.jp/
- 其他物种
- JGI https://phytozome.jgi.doe.gov/pz/portal.html#
- NCBI
- GEO https://www.ncbi.nlm.nih.gov/geo/
- SRA https://www.ncbi.nlm.nih.gov/sra
- 其他
- ENSEMBL
- 在线学习 https://www.ebi.ac.uk/training/online/
- EnsemblPlants http://plants.ensembl.org/index.htmls
- 蛋白
- EBI http://www.ebi.ac.uk/services/proteins
- pfam http://pfam.xfam.org/
- uniprot http://www.uniprot.org/
- GPOFILER http://biit.cs.ut.ee/gprofiler/index.cgi
- GO http://www.geneontology.org/
- 生物信息工具查询网站
- https://bioinformatics.ca/links_directory/
- https://omictools.com/
- https://www.atum.bio/resources/bioinformatics-tools
- https://wiki2.org/en/List_of_RNA-Seq_bioinformatics_tools
5. 生物信息统计入门基础
- 基本概念
- 统计方法
- 实现方法
- 结果可视化
- 相关软件算法实现
6. 编程基础
- Linux shell
- R http://www.sthda.com/english/
- Python
- 机器学习
- 在线课程
- 生信编程题目练习 http://rosalind.info/problems/locations/
- 慕课网 http://www.imooc.com/
7. 生物学基础知识
- 基因组相关
- 转录组相关
- 表观组相关
- 在线课程网站
- coursera https://www.coursera.org/
- edX https://courses.edx.org/
8. 思维导图