第3章 基础转录机器
3.1 核心启动子
-
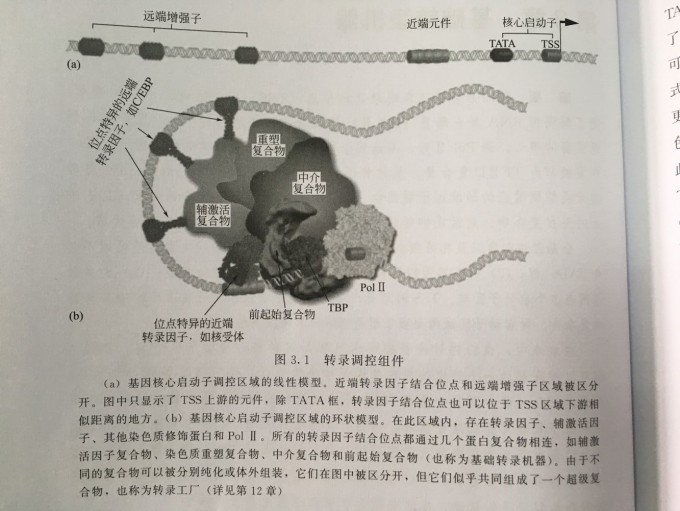
转录因子结合到远端增强子上,招募大量辅激活因子,在成环的条件下,对靶基因的转录活动产生影响。
-
通过这种方式,转录因子可以接触到核心启动子区域,在核心启动子区域,PolⅡ与大量的通用转录因子组装在一起。
-
核心启动子位于TSS上下游50bp的一段基因组区域。
3.2 TATA框
-
在核心启动子上,了解最清楚的是TATA框,它是TBP的结合位点。
-
TATA框是位点特异性转录因子结合的一段DNA基序,TATA的名字是其共有序列TATAWADR的简称。
-
共有序列通常用于表示已知转录因子结合位点的属性。
3.3 核心启动子元件
-
TSS区的核心启动子DNA上经常缺少核小体,即核心启动子区大多数是易接近的。
-
在核心启动子区有不同类型的通用转录因子结合位点。
-
起始子元件Inr在功能上类似于TATA框。
-
在TATA框缺失的启动子中,常常发现DPE(下游核心启动子元件)的存在,其位于TSS下游约30bp,DPE与Inr结合作用,指导特定的转录起始。
-
在管家基因中,经常发现核心启动子区既缺少TATA框又缺少Inr,但包含多个高C含量的结合位点。
-
在同源基因中,核心启动子的序列元件一般比较保守。
-
人类基因组启动子序列最主要的特征:只有大约60%的人类基因的核心启动子区靠近CpG岛。
3.4 基因组范围内识别核心启动子的方法
-
高通量测序可用于mRNA 5’端或活性TSS的定位。
-
这些方法要求高度的严谨性,需要在测序前捕获全长mRNA分子的cDNA拷贝。
-
ChIP-seq分析能够识别结合于活性基因启动子上的PolⅡ。
3.4 补充延伸–RNA-seq
转录组是指细胞在特定的发育阶段或生理条件下,其转录本的完整集合。在RNA-seq中,RNA分子[总RNA或某些类型的RNA,如ploy(A)+ mRNA]反转录成一个cDNA片段文库,并在cDNA的一端或两端加上接头。不管有没有被扩增,每个分子都通过高通量的方法被测序。
3.5 中介复合物
-
调控信号是通过中介复合物呈递的。
-
中介复合物是一个信号归类中心,参与几乎所有基因的转录调控,并且介导大多数转录因子的作用。
-
中介复合物也可以直接调节染色质重塑和基础转录机器之间的转换。
第四章 转录因子
-
根据转录因子表达的部位可以分为广泛性表达和组织特异性表达。
-
根据表达方式可以分为组成型表达(持续性激活)和调控型表达(受到胞内或胞外信号调控)。
4.1 位点特异性转录因子
-
位点特异性转录因子对于对于决定一个基因是否表达以及表达的程度非常关键;
-
转录可被具有阻遏功能的转录因子下调;
-
位点特异性转录因子在DNA上的结合位点可以离TSS区具有任意的距离。
4.2 转录因子结构域
-
典型转录因子的结构域:与DNA结合的结构域(DBD)、与其他转录因子形成二聚体的结构域、借助辅因子与基础转录机器相互作用的结构域。
-
根据DBD对TF进行分类:(1)zinc finger (2)helix-turn-helix (3)homeodomain。
-
结构域中用于蛋白质与蛋白质互作的基序主要有三种:(1)leucine zipper (2)bHLH (3)β-scaffold factors with minor groove contacts。
4.4 生物信息学方法识别转录因子结合位点
-
可利用计算机识别的转录调控元件:①含有TSS区的核心启动子 ②较远的增强子和LCR ③沉默子、绝缘子和基质附着位点。
-
徒劳定理:任何计算机筛选预测出的转录因子结合位点数较实际超过1000倍。
-
复合物形成中涉及的蛋白质-蛋白质相互作用越多,利用生物信息学的方法来识别转录因子的体内结合位点就会越困难。
4.5 转录因子表达谱
-
人类的转录因子数量在1400-1900之间,占所有编码蛋白质基因的7%-9%。
-
与大多数参与信号转导的蛋白质一样,转录因子的表达量不是很高。
-
转录因子表达的两种极端情况:它们或者只在一两个组织中受到非常严格限制的表达,或者在几乎所有组织中都广泛表达。
-
大多数转录因子都可归于一个特定家族。
4.6 转录因子的分类

第三、四章重要概念
第三章重要概念
-
核心启动子是位于TSS +/- 50bp的基因组区域。为组装基础转录机器,并将PolⅡ置于正确的位置以起始转录,核心启动子区包含所有必要的转录因子的结合位点。
-
只有10-20%的哺乳动物核心启动子带有一个功能性TATA框。核心启动子的替代性结合位点是Inr、DPE和BRE位点。大约有60%的人类核心启动子位置靠近CpG岛,并可能受转录因子SP1指导。
-
ChIP-seq分析能够识别结合于活跃基因启动子区域的或与增强子区域也具有联系的PolⅡ。ChIP-seq的结果表明,远端结合的转录因子通过蛋白质-蛋白质之间的相互作用与基础转录机器连接。
-
锋利核心启动子更有可能包含TATA框,而广泛核心启动子通常接近CpG岛。锋利启动子(即含有TSS的启动子)似乎优先用于特定组织的基因表达,而广泛启动子(即启动子区域缺乏一个明显的TSS)一般与普通表达的基因相关。
-
大多数人类蛋白质编码基因有多个核心启动子区域。这些可变启动子通常存在于不同的组织中,以合成不同的蛋白质产物,这导致了人类蛋白质组的复杂性。
-
中介复合物的头部和中部模块与基础转录机器相互作用,而尾部亚基与各种各样的转录因子相互作用。激酶模块与PolⅡ相互作用,激酶模块缺失时,转录抑制。中介复合物可以感受并收集大量的信号,并将正确的输出信号传递给转录机器。
第四章重要概念
-
一个基因的转录水平可通过位点特异性转录因子的激活显著提高。
-
转录因子结合位点和TSS区的线性距离不是关键的。
-
超过150种转录因子,包括原癌基因产物、肿瘤抑制蛋白等,与300多种疾病直接相关。
-
一个典型的转录因子包括特征性的DBD区和与其他转录因子和辅因子之间发生蛋白质-蛋白质相互作用的部位。
-
转录因子DBD区通过识别DNA分子大沟内的特异性碱基位点与基因组DNA发生相互作用。但是转录因子单体识别位点不超过6bp。
-
计算机筛选很容易过度预测结合位点,因此,为了提供更加可靠的预测,生物信息学方法需要实验数据如ChIP-seq获得的数据来迅训练。
-
转录因子在不同细胞型中的数量变化很大,从每个细胞约100个,仅调控几个目标基因的高度特异性转录因子,到每个细胞超过100000个,参与调控大多数基因的遍在转录因子。
-
转录因子的表达倾向于两种极端情况:它们或者只在一两个组织中受到非常严格限制的表达,或者在几乎所有组织中都广泛表达。