本文参考了部分文献及《植物研究进展》、《植物科学最前沿》公众号的三篇文章。
本文主要以本人所做的水稻NBS-LRR基因家族的分析为例。
1. 基因家族的鉴定
-
首先根据pfam数据库中NBS-LRR基因家族的的保守结构域NBS(PF00931)信息,利用HMMER软件构建种子序列,并对日本晴全基因组蛋白质序列进行搜索,如果存在该结构域,则判定为NBS-LRR候选基因;
-
接着,使用PFAM、PANTHER、SUPERFAMILY等数据库对候选基因进行进一步确认;
-
需要注意的是,对于部分在Pfam数据库里没有被收录的基因家族,我们只能在模式植物,如拟南芥中搜集已知的基因家族成员序列,然后在自己研究的物种中进行序列比对,从而对基因家族成员进行鉴定。(摘录于植物研究进展)
2. 进行染色体定位和基因簇划分
-
首先提取候选基因集的gene locus信息,使用MapInspect等绘图软件绘制其染色体定位信息;
-
gene cluster的划分原则:two or more NBS-LRR genes locus resided within 200 kb。
3. 构建系统进化树和加倍复制分析
-
系统进化树的绘制可以使用MEGA7.0软件,为了方便对基因进行分类,应该选取日本晴中的NBS-LRR家族及拟南芥中的NBS-LRR家族一起来分析,图例如下:
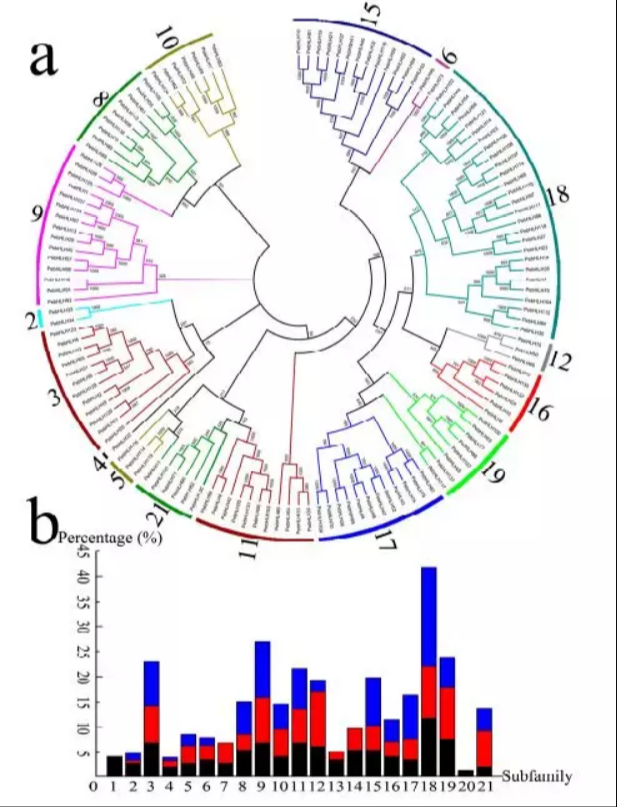
-
利用MCScanX软件可以做共线性分析,如下:
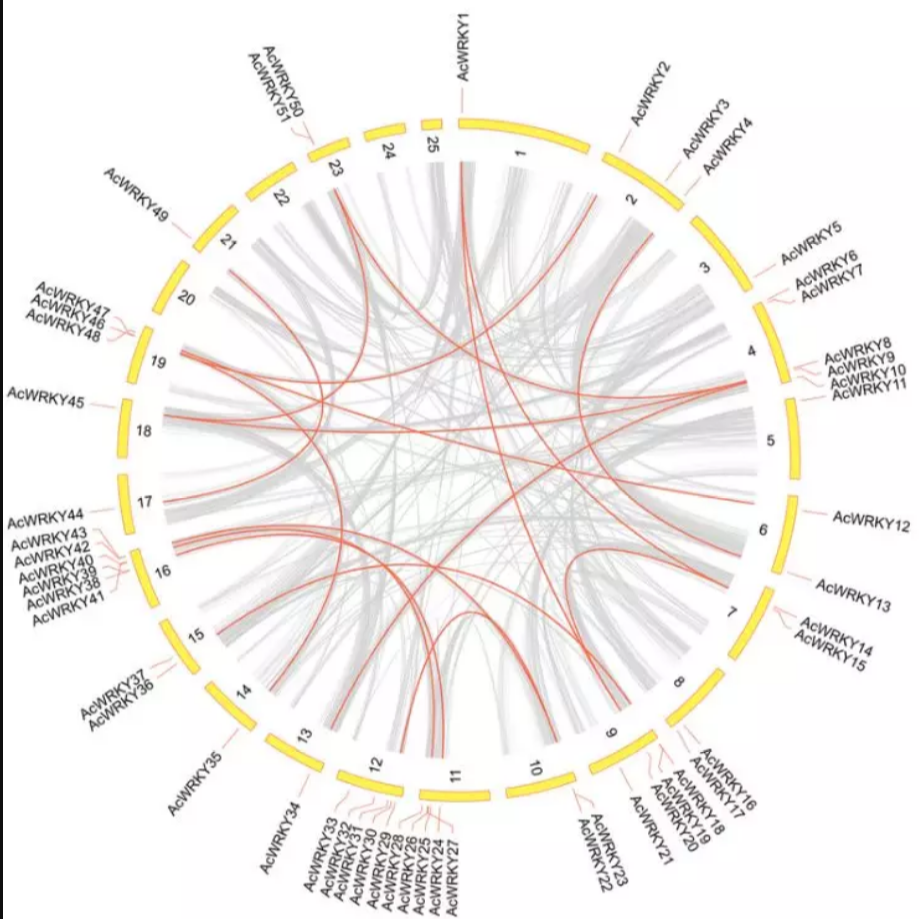
4. 基因结构和motif分析
利用MEME绘制基因的外显子和内含子位置信息,以及基因序列上的motif信息,并将这些基因结构信息按照系统进化树的顺序进行展示。
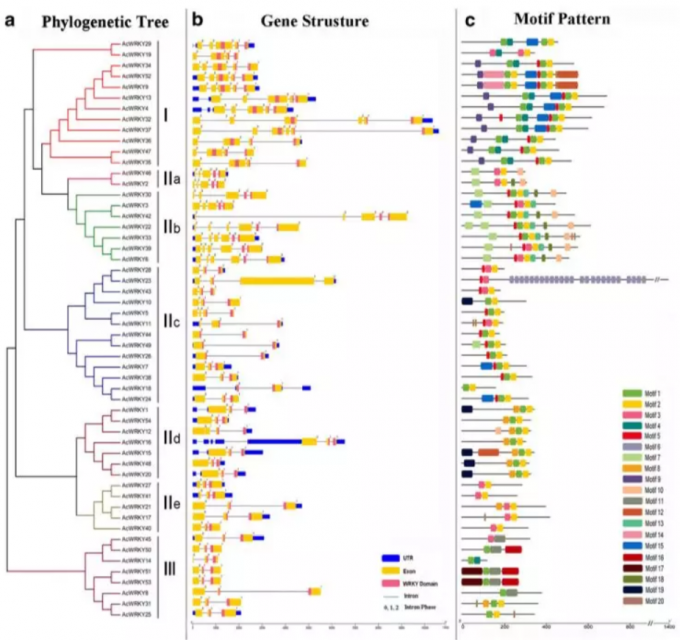
5. 水稻和拟南芥蛋白家族基因的同源性分析
拟南芥中NBS-LRR蛋白功能研究较为完善,进行水稻与拟南芥间NBS-LRR蛋白同源性分析有助于解析水稻中NBS-LRR蛋白的功能,具体信息见下图:
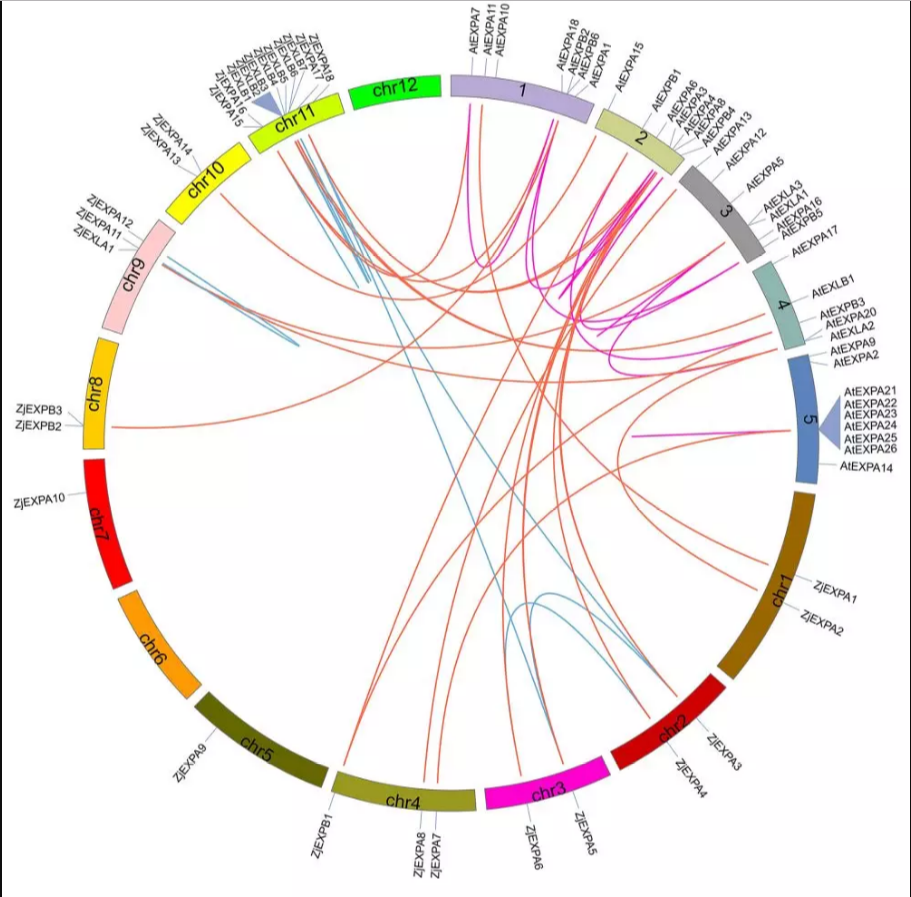
6. 蛋白家族启动子区域顺式作用元件分析
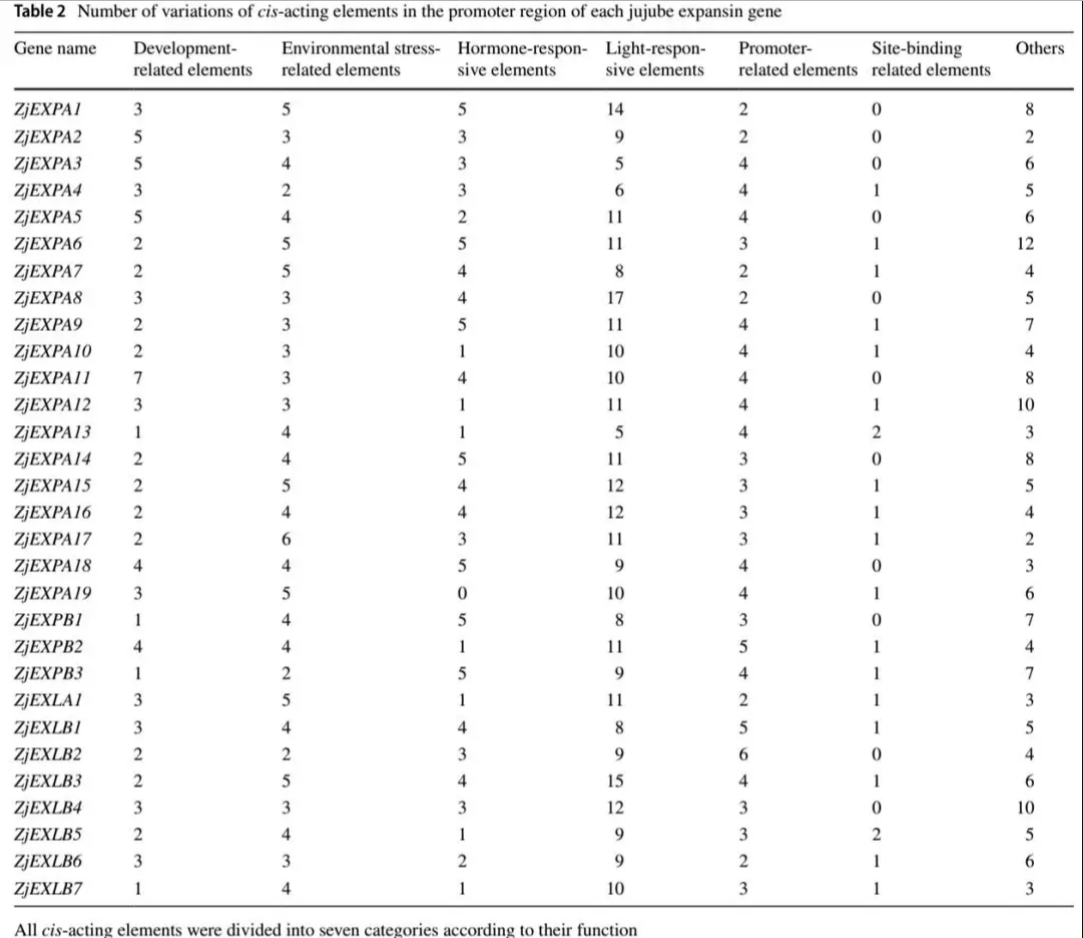
具体可以使用PLANTCARE来挖掘基因中的顺势元件。
7. 基因多重序列比对、GO注释
-
多重序列比对可以使用CLUSTAL OMEGA软件;
-
GO注释可以使用水稻基因组注释文件或WEGO在线。
8. 基因家族启动子及表达模式分析(以毛竹bHLH为例)
由于那篇文章研究的基因家族是转录因子,所以可以对家族内基因启动子区域进行分析,这也是本文分析比较具有亮点的部分;bHLH基因家族所含有的顺式作用元件共可划分为3类:一是光周期响应原件;二是植物生长发育响应原件;三是生物/非生物胁迫响应原件。
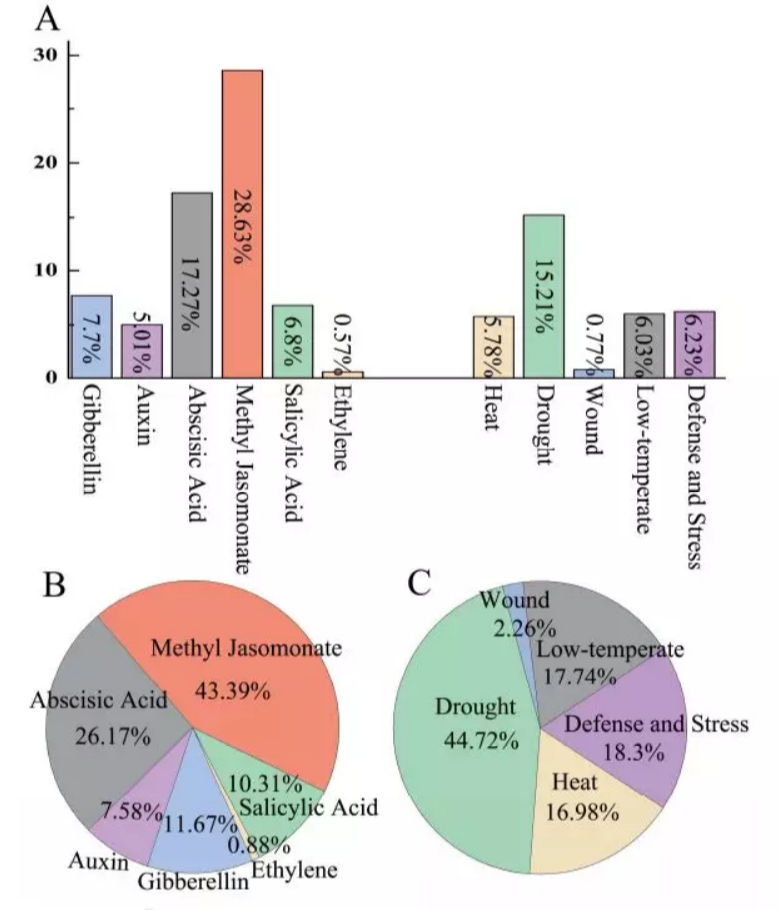
9. 基因在不同处理下的差异表达分析及可视化(和基因表达量数据的结合)
10. qRT-PCR检测胁迫环境下NBS-LRR基因表达模式变化
基因家族的主体分析内容完成之后,还可以结合qPCR等基因表达数据进行进一步挖掘。
