参考资料:橙子牛奶糖的博客
1. 数据预处理(DNA genotyping、Quality control、imputation)
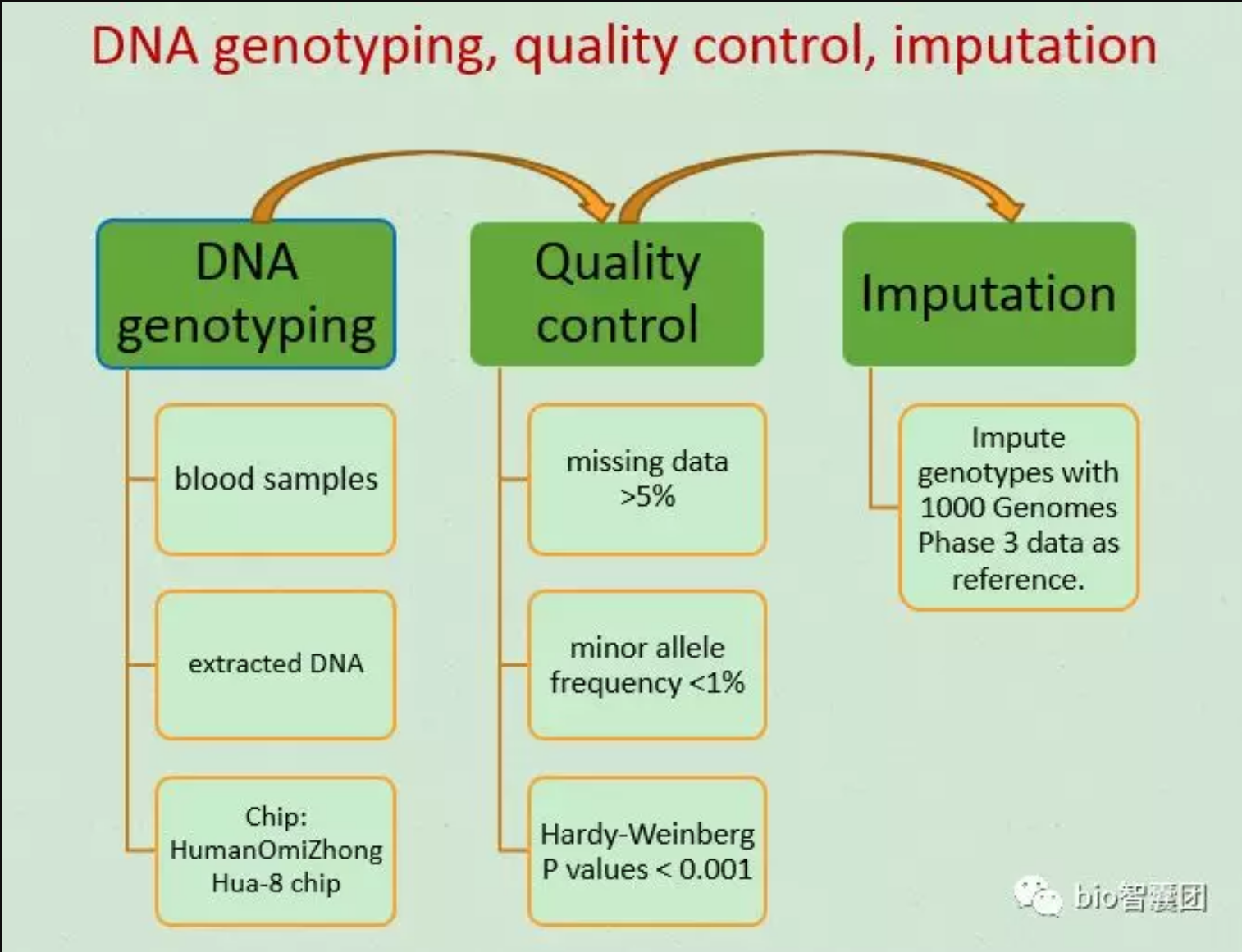
QC的工作可以用PLINK完成,Imputation的工作可以用IMPUTE2完成。
2. 表型数据统计分析
-
逻辑回归(表型数据为二元)
-
线性回归(表型数据为连续性变量)
-
表型数据正态分析(如果不是正态分布,需转换处理为正态分布)
-
表型数据均值、中值、最大值、最小值
-
影响因子对表型的影响分析
3. 画曼哈顿图(GWAS)和QQ plot图
3.1 准备plink文件
3.1.1 准备PED文件
PED文件是空格(空格或制表符)分隔的文件,共有六列,内容如下:
- Family ID
- Individual ID
- Paternal ID
- Maternal ID
- Sex (1=male; 2=female; other=unknown)
- Phenotype

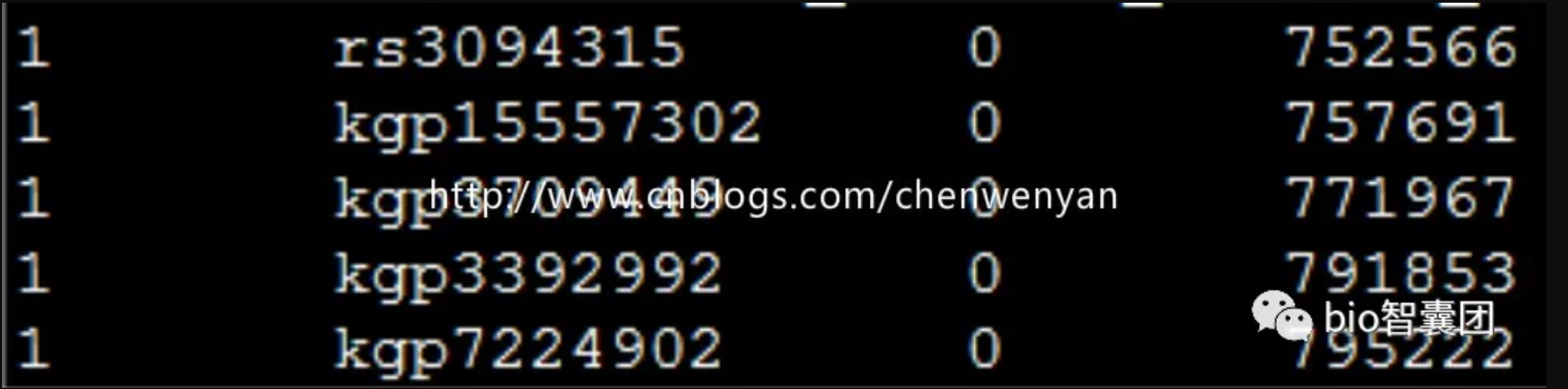
3.1.2 准备MAP文件
MAP文件有四列,四列内容如下:
- chromosome (1-22, X, Y or 0 if unplaced)
- rs# or snp identifier
- Genetic distance (morgans)
- Base-pair position (bp units)
** 3.1.3 生成bed、fam、bim文件**
在plink中输入命令:plink –file mydata –out mydata –make-bed
plink指的是plink软件,如果软件安装在某个指定的路径的话,前面还要加上路径,比如安装在路径为/your/pathway/的文件夹下,则命令应该为“/your/pathway/plink –file mydata –out mydata –make-bed”
mydata指的是1和2生成的PED和MAP文件名,不需要写.ped和.map后缀
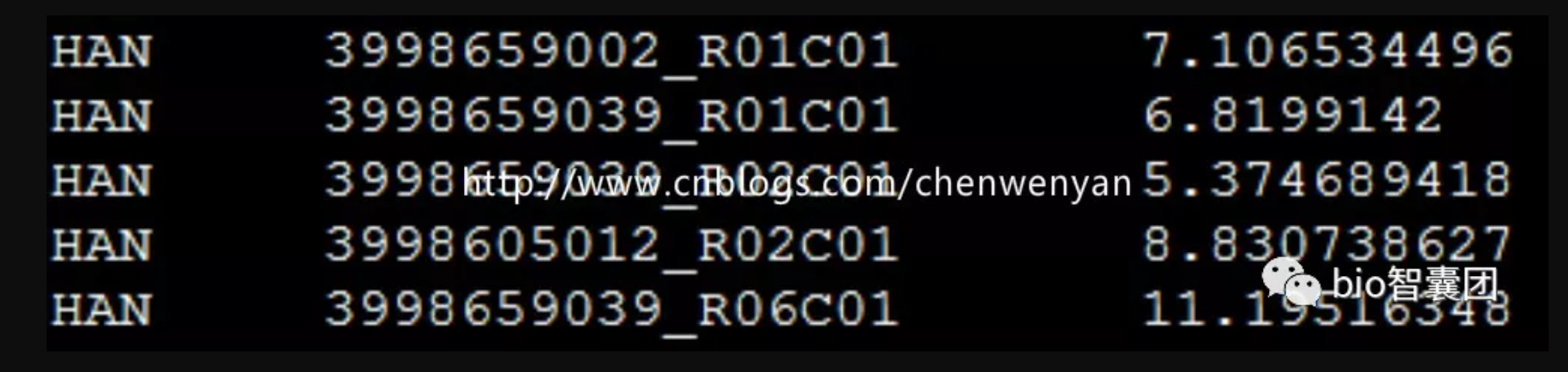
3.2 准备表型文件(Alternate phenotype files)
一般表型文件为txt格式,表型文件有三列,分别为:
- Family ID
- Individual ID
- Phenotype
假如有多种表型,第一列和第二列还是Family ID、Individual ID,第三列及以后的每列都是表型,例如以下:
- Family ID
- Individual ID
- Phenotype A
- Phenotype B
- Phenotype C
- Phenotype D
- Phenotype E
- ……
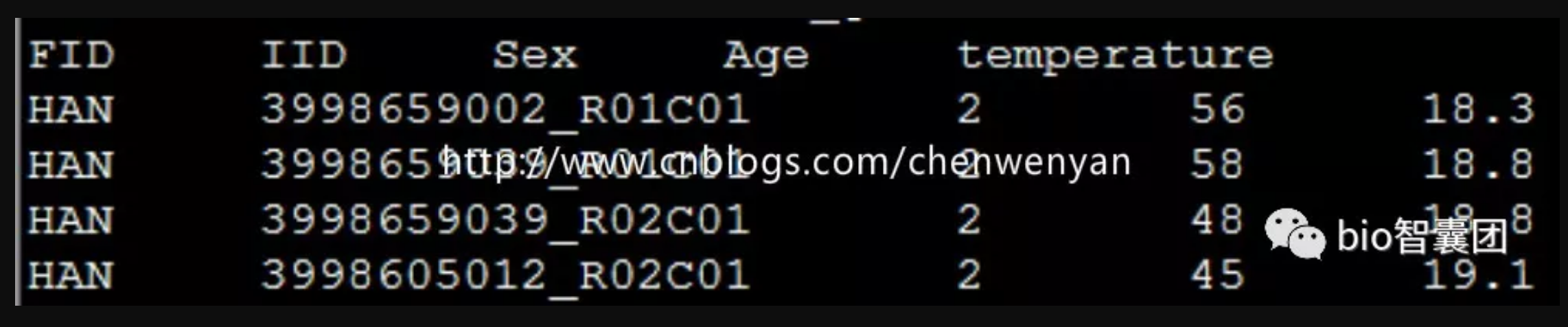
3.3 准备协变量文件(Covariate files)
协变量文件同表型文件类似,第一列和第二列是Family ID、Individual ID,第三列及以后的每列都是协变量:
- Family ID
- Individual ID
- Covariate A
- Covariate B
- Covariate C
- Covariate D
- Covariate E
- ……
3.4 使用PLINK进行表型和基因型以及协变量的关联分析
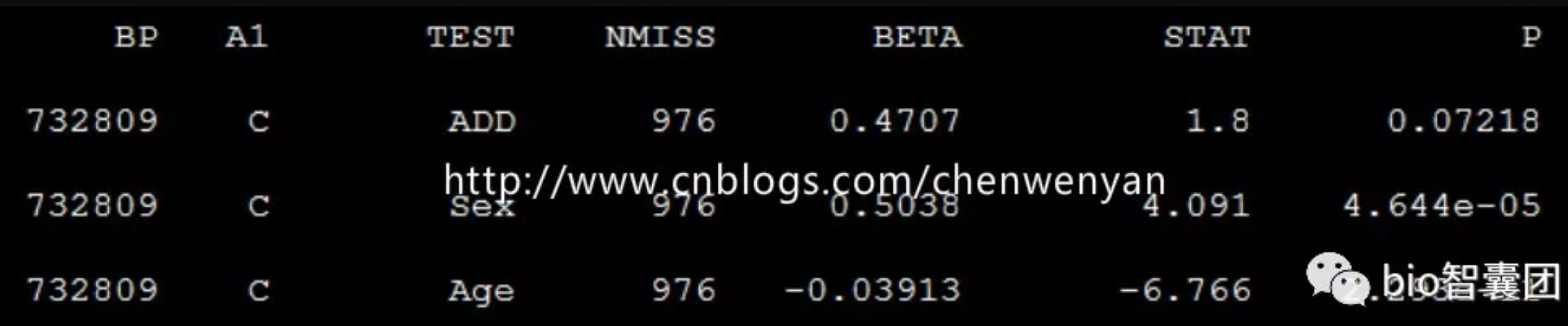
命令如下:plink –bfile mydata –linear –pheno pheno.txt –mpheno 1 –covar covar.txt –covar-number 1,2,3 –out mydata –noweb
生成的文件为mydata.assoc.linear
注:“mydata”:mydata文件不需要后缀
“–mpheno 1”指的是表型文件的第三列(即第一个表型)
“–covar-number 1,2,3”指的是协变量文件的第三列、第四列、第五列(即第一个、第二个、第三个协变量)
“–linear”指的是用的连续型线性回归,如果表型为二项式(即0、1)类型,则用“–logistic”
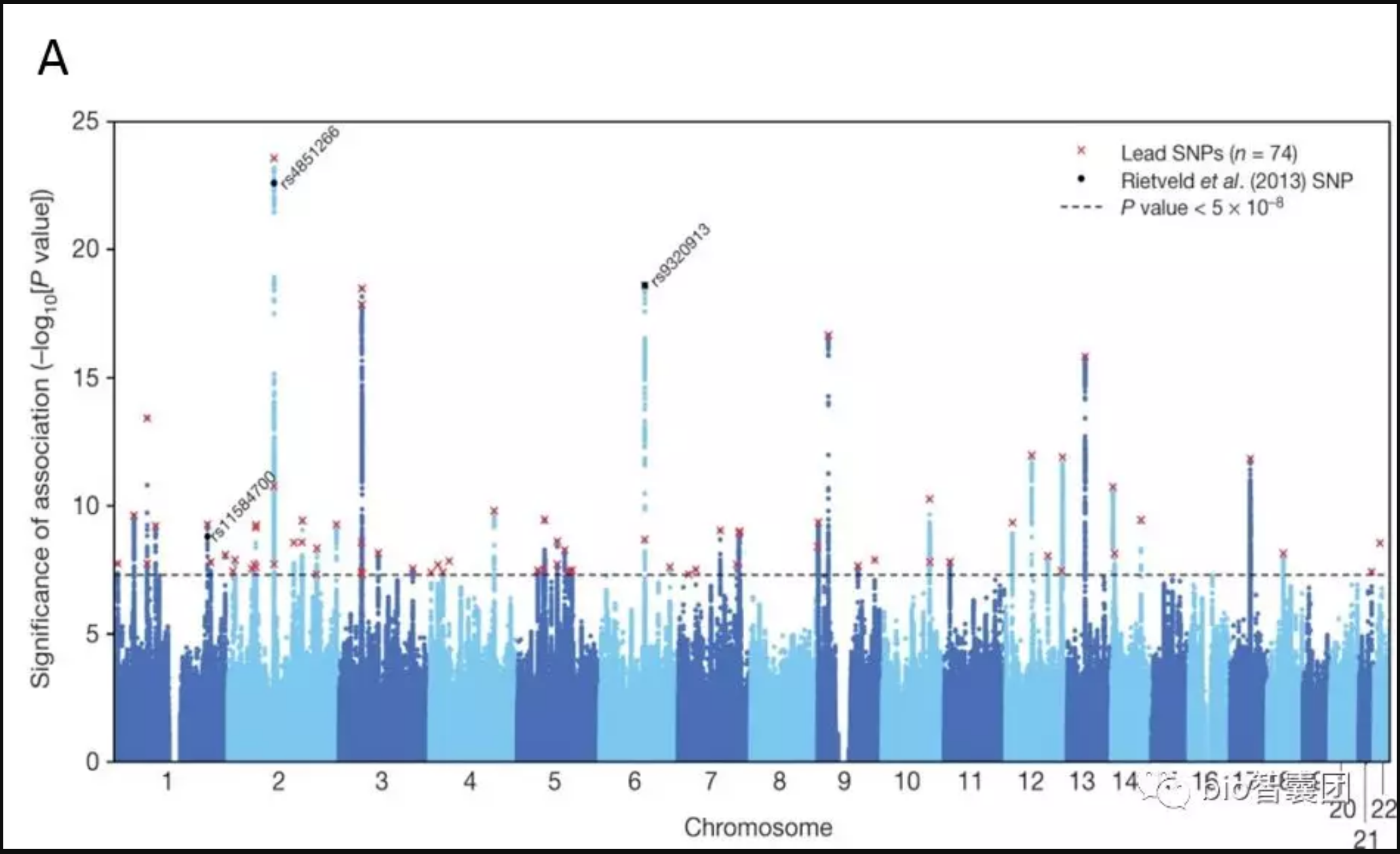
3.5 画曼哈顿图
安装R语言的CpGassoc包,其中的manhattan(),即可画曼哈顿图。
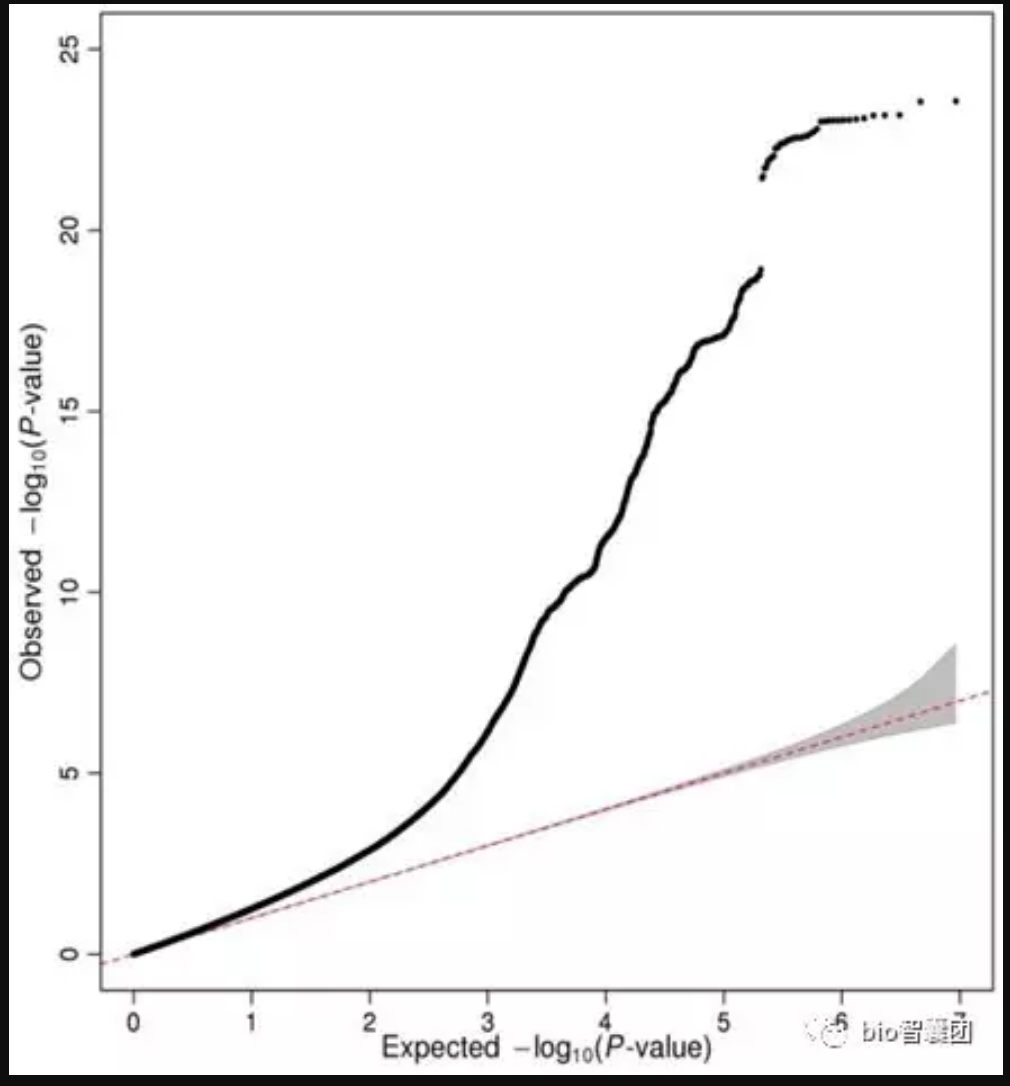
3.6 画QQ plot图
R语言中的 qqnorm() 和 qqplot() 包提供了QQ plot的画法,具体自行搜索用法。
4. GWAS进阶分析
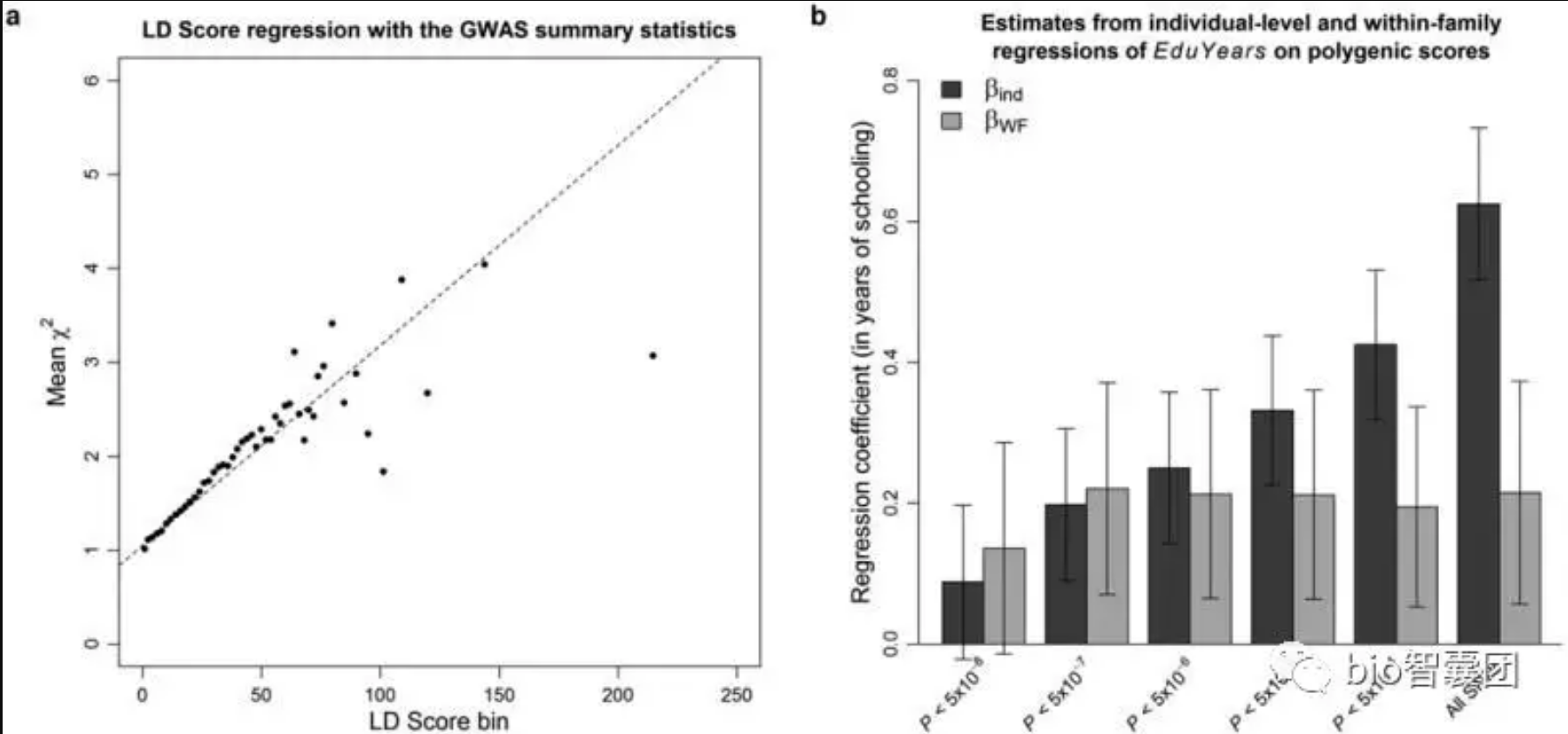
4.1 群体分层分析(Population Stratification)
如果研究的群体是混合群体,遗传异质性高,存在群体分层现象,易造成实验的误差或者检测出假阳性位点。因此检测群体分层对效应值的影响是非常必要的。
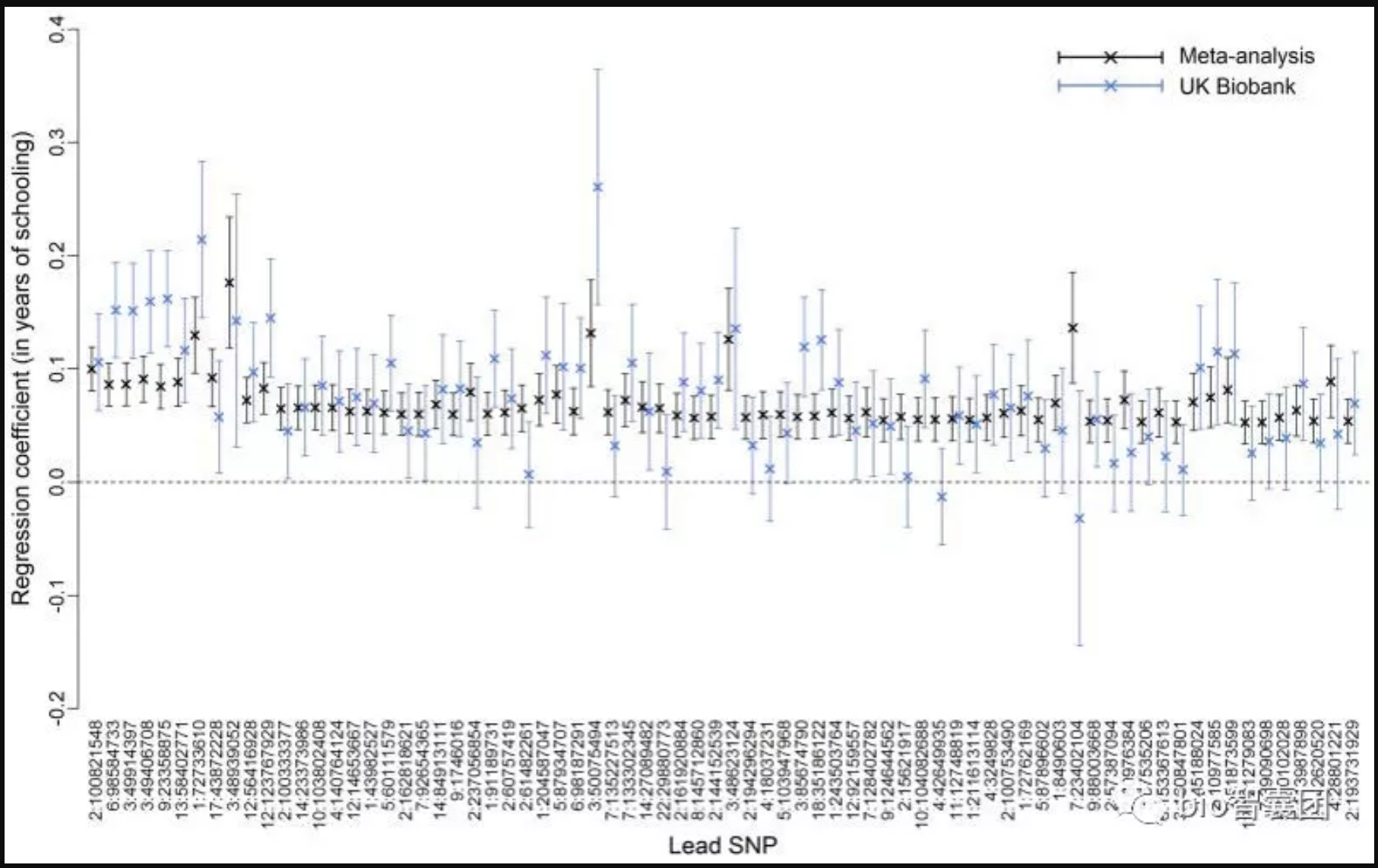
4.2 不同群体重复验证分析(Replication)
4.3 区域关联图(Regional association plots)
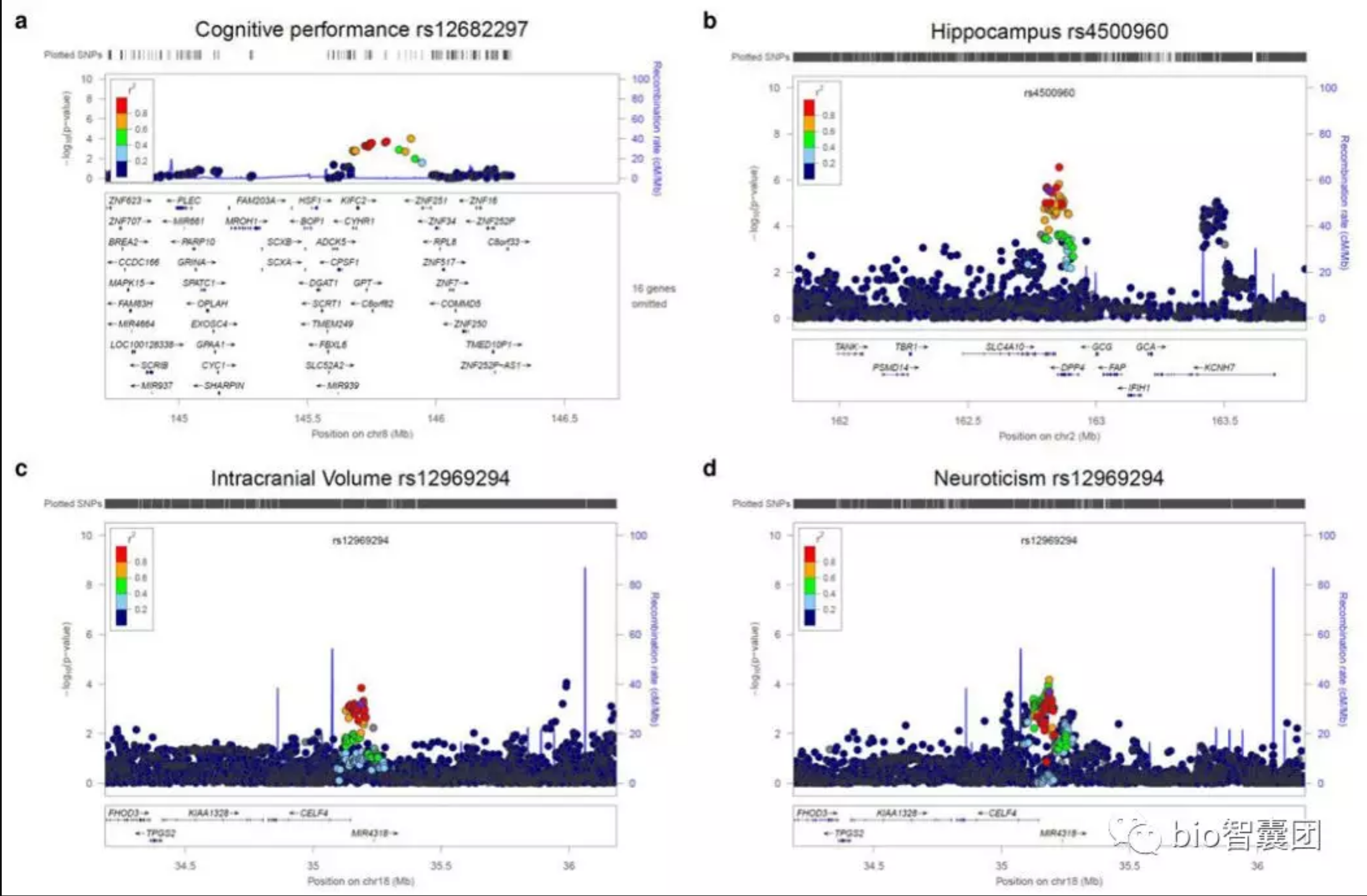
用LocusZoom(http://csg.sph.umich.edu/locuszoom/)画出来的
4.4 相似条件分析(Approximate conditional analysis)
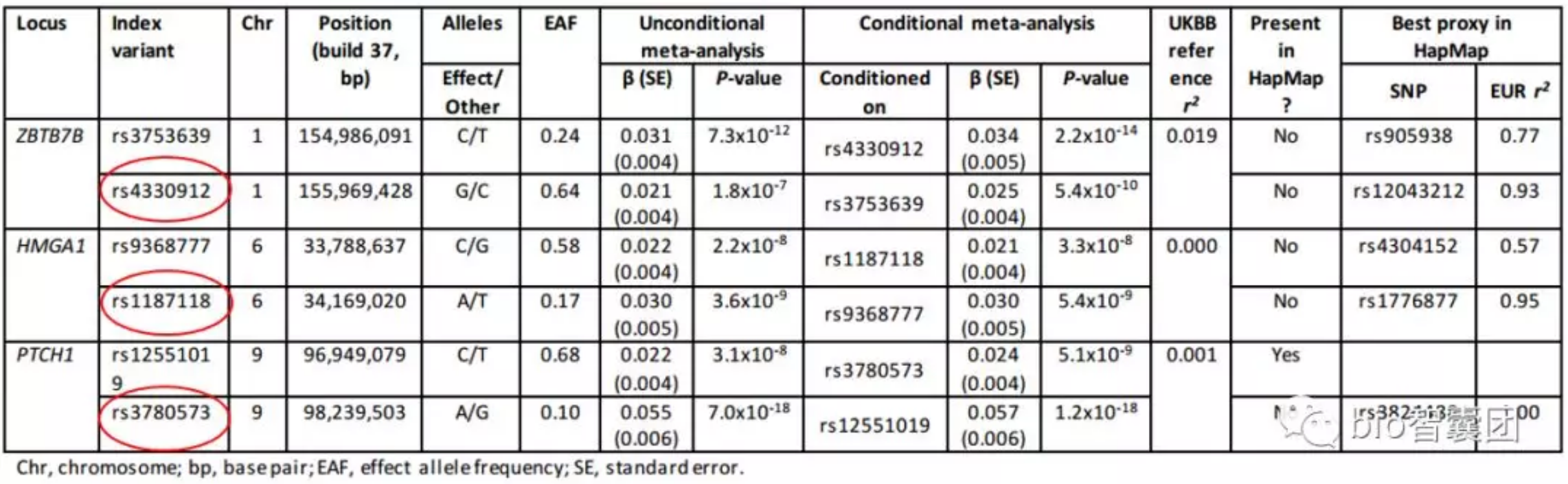
相似条件分析的目的是,去掉lead SNPs后,再跑一次GWAS关联分析,以此找到更多有强关联的信号。
4.5 连锁不平衡得分评估表型间遗传相关性(Linkage-disequilibrium score regression)
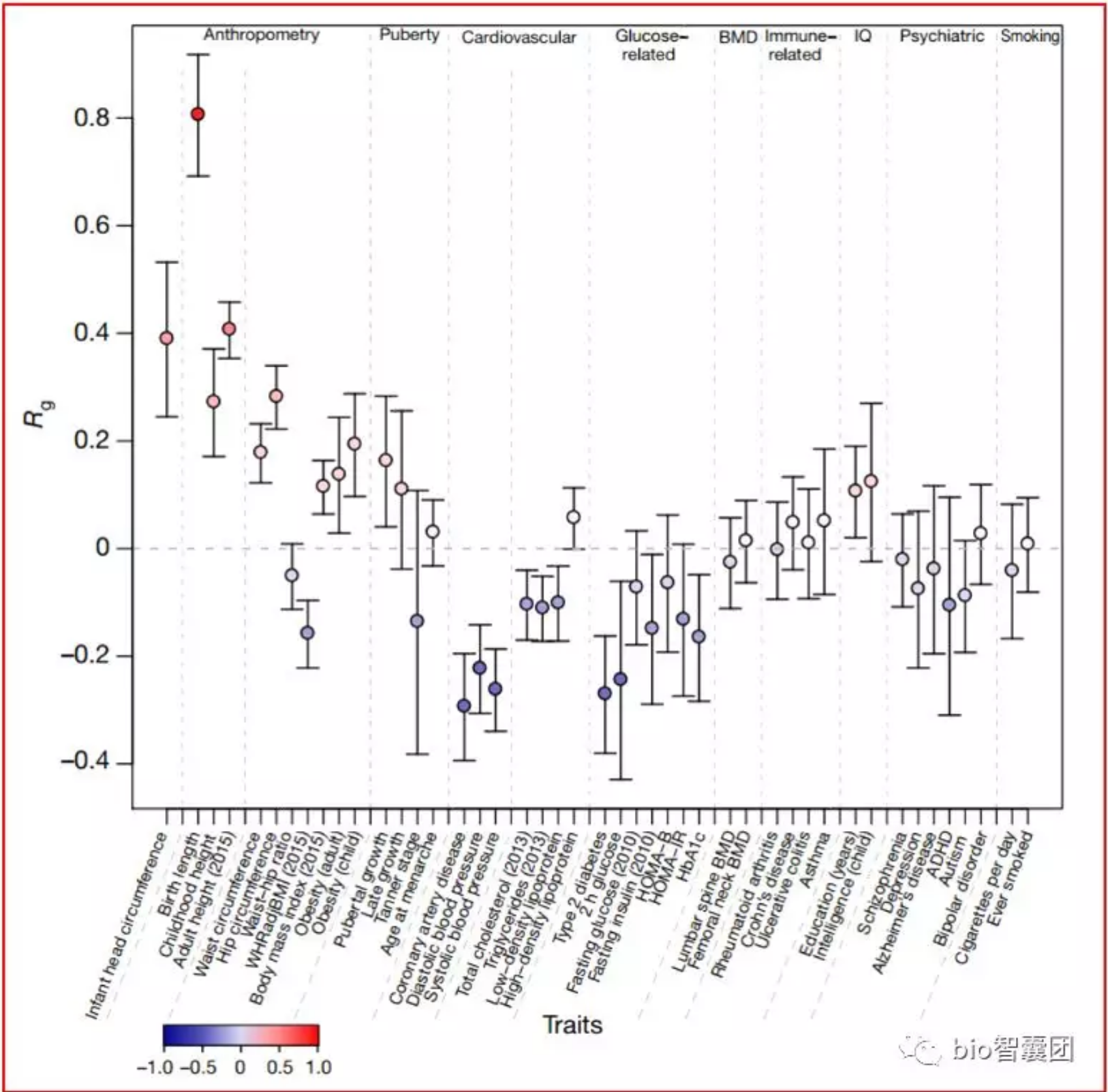
遗传相关性 genetic correlation, 这是指在杂种群体表型间的相关性中,由基因型所产生的相关性。遗传相关是仅由遗传原因引起的相关。
4.6 基因富集分析(Gene setenrichment analysis)
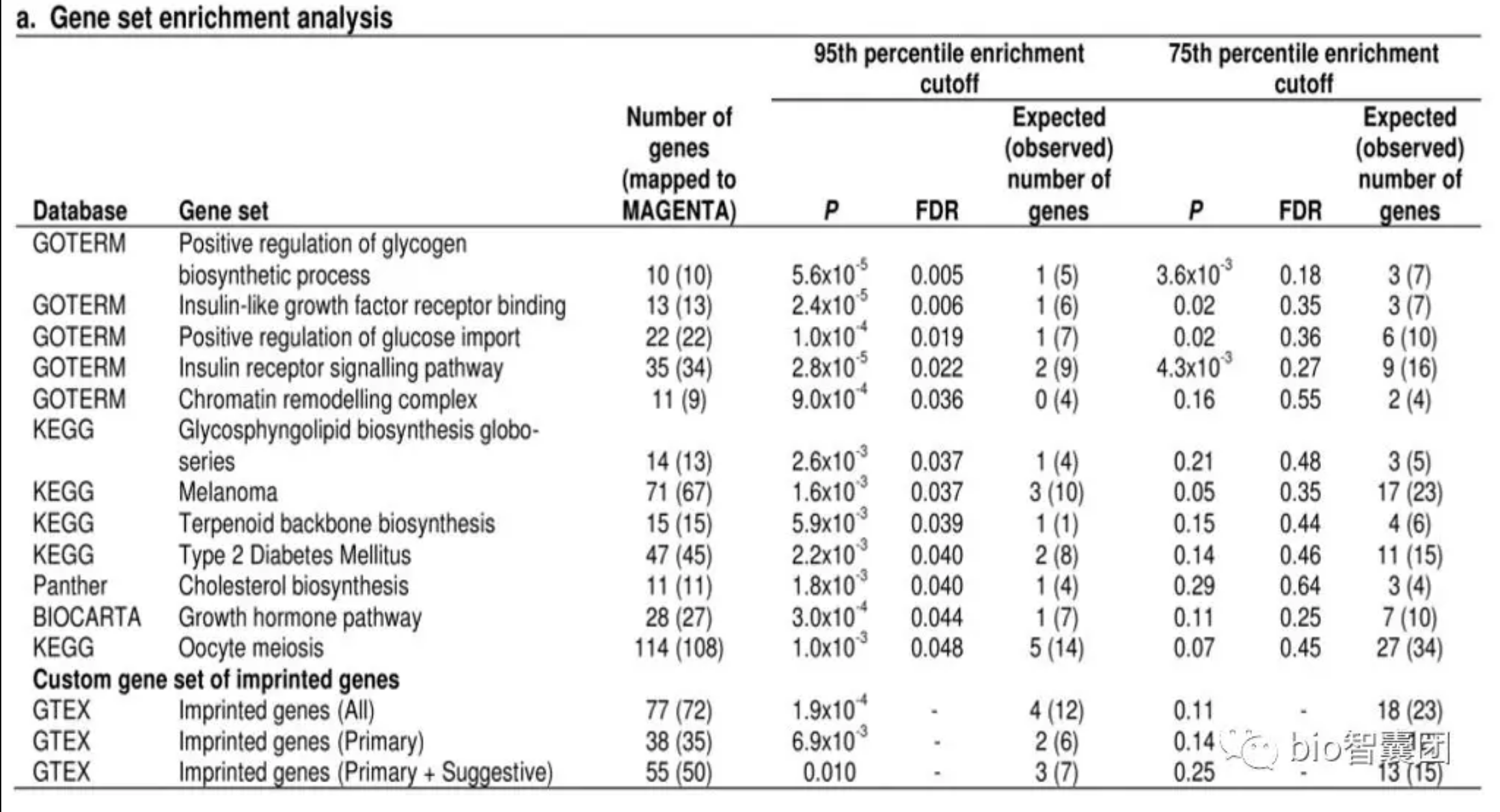
从文献,GTEX、 GEUVADIS等据数据找到与 Lead SNPs 关系很大的基因、变异等,以及与 Lead SNPs 连锁不平衡分析r2大于0.8的SNP,则这些SNP的基因可以作为候选基因。汇总了所有的候选基因后,在GOTERM、KEGG、Panther等数据库分析这些候选基因的富集分析。
4.7 层次聚类分析(Hierarchicalclustering)
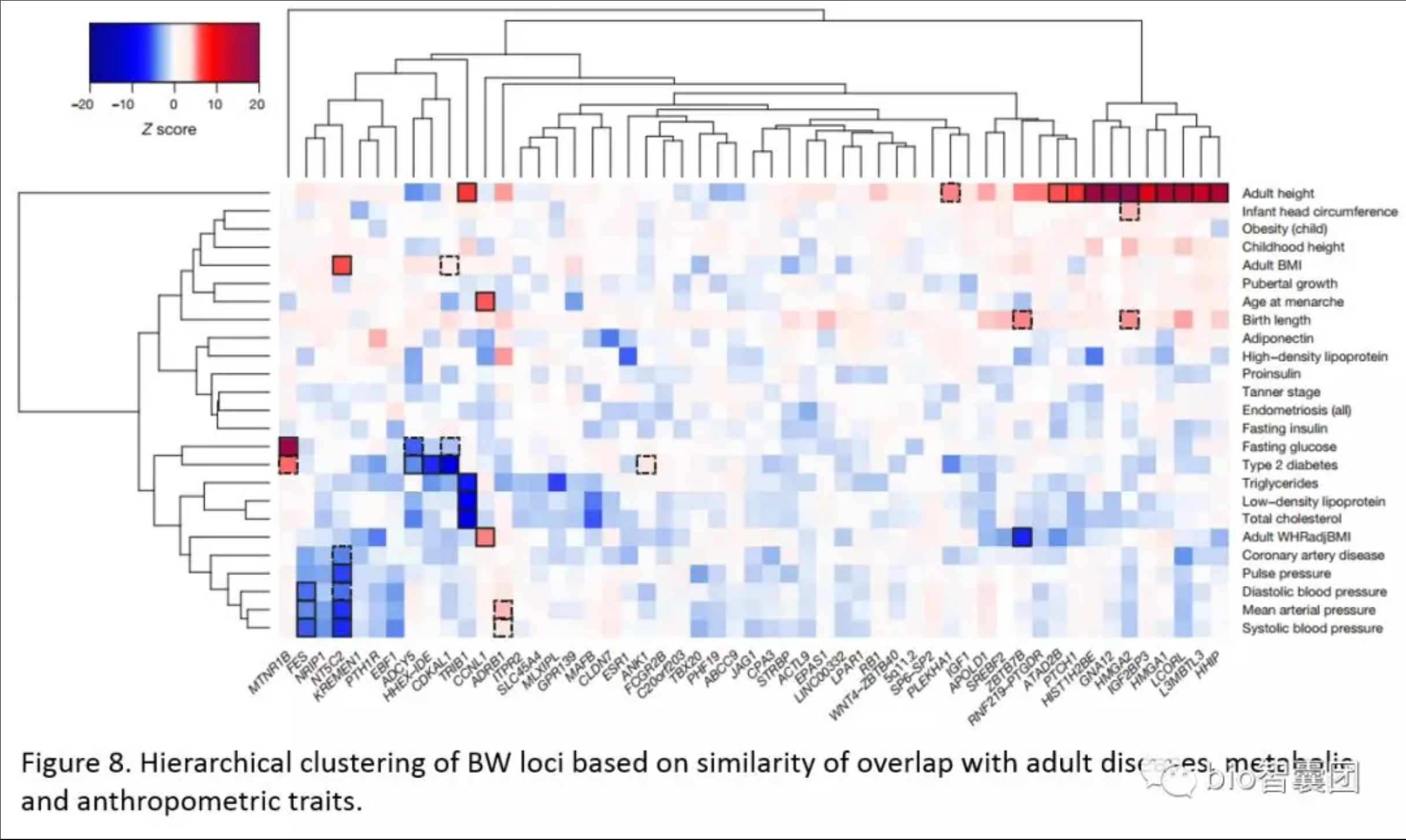
层次聚类分析的作用是更进一步看出研究的表型相关的 lead SNPs与其他表型的相关性.
4.8 蛋白质-蛋白质互作网络分析(Protein–proteininteraction network analyses)

蛋白质互作网络的目的是看哪些蛋白共同调控了表型
4.9 接触点分析(Point ofcontact analyses)
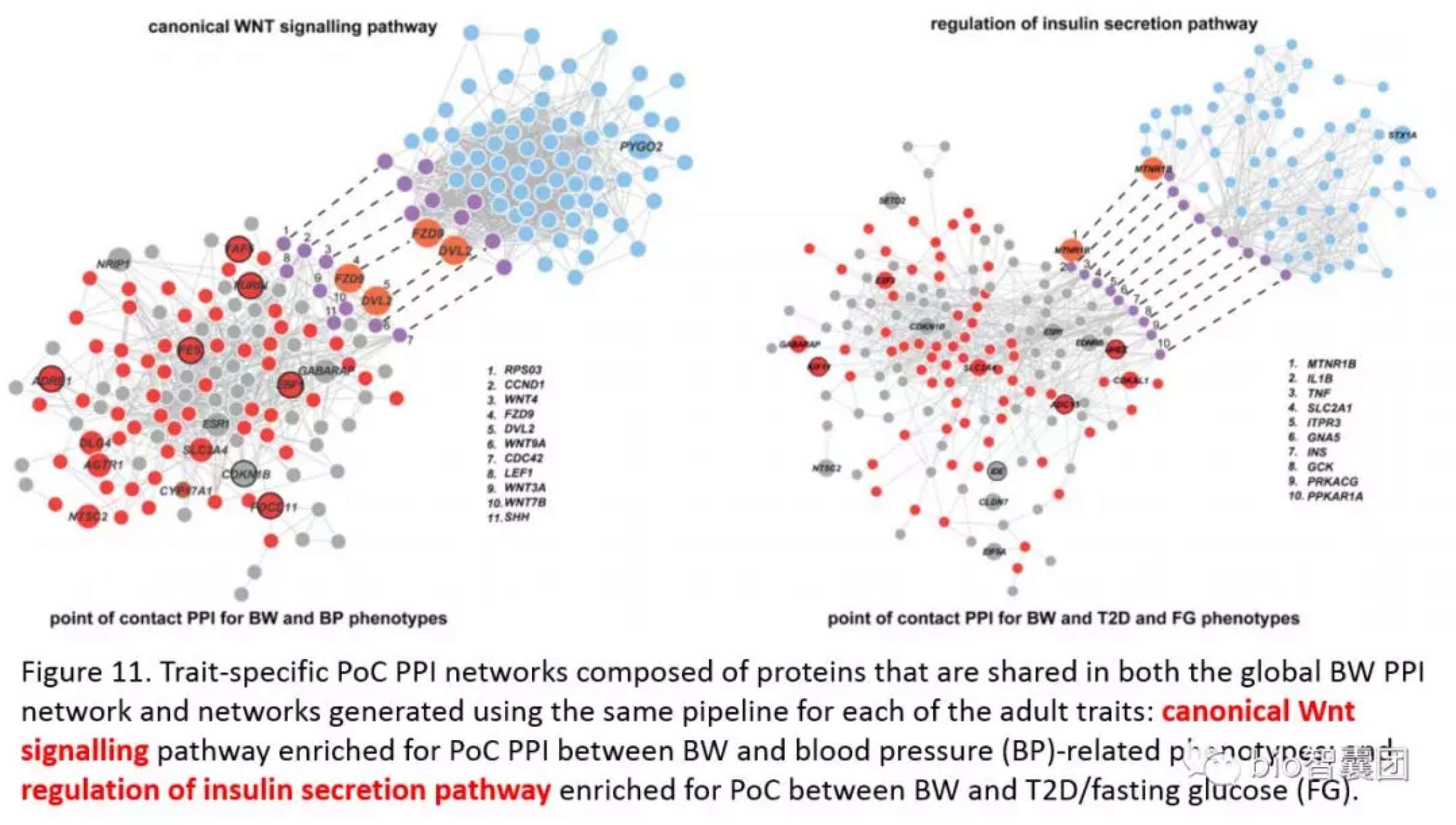
Point of contact analyses的作用是哪些位点导致了表型间有相关性