资料来源:公众号:育种数据分析之放飞自我
1.前言
-
之前的教程中,我们使用的是别人模拟的数据,数据类型是二分类数据,这里我们模拟一个数量性状的连续性状,做GWAS更有代表性。
-
我们先从没有协变量的一般线性模型(LM)开始,然后加入数据类型的协变量,然后加入因子类型的协变量(这里需要进行虚拟变量的转化),然后将数值协变量和因子变量放在一起作为协变量,然后将PCA的值作为协变量加进去,这样一般线性模型分析完成。
-
最后我们使用混合线性模型(LMM),不考虑协变量,然后加入协变量,然后将PCA加入协变量。
本篇主要是介绍一般线性模型(LM)中的assoc和linear两个参数,不考虑协变量,然后将结果与R语言编程结果比较。
2.模拟的数据
模拟数据使用QMSim软件,模拟的数据如下:
这里b.map和b.ped是plink格式的数据,cov.txt为协变量,phe.txt为观测值。
b.map数据:
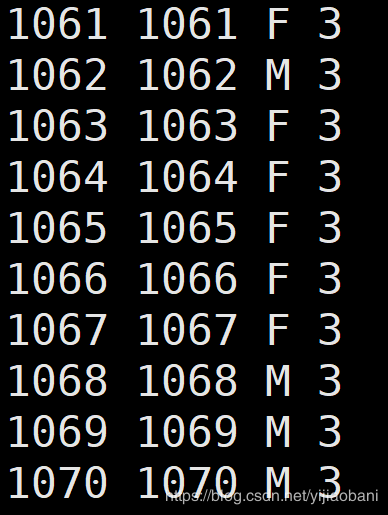
b.ped数据:
cov.txt 协变量数据:
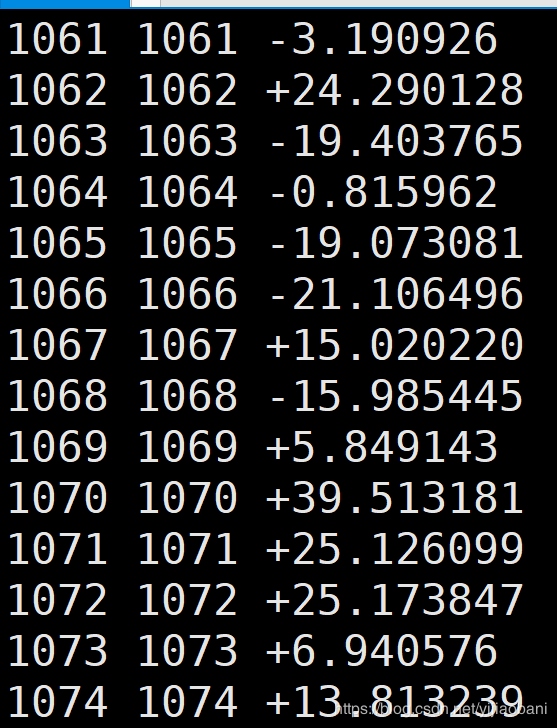
phe.txt表型数据:
3. plink利用assoc进行LM的GWAS分析
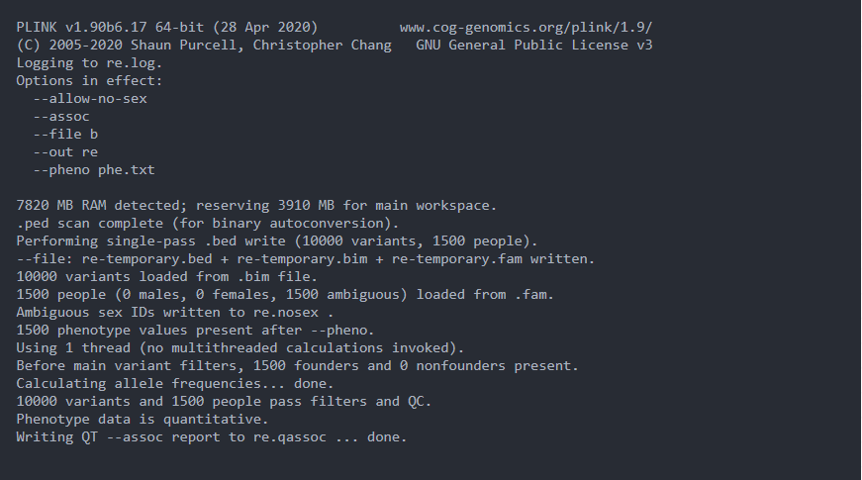
plink --file b --pheno phe.txt --assoc --out re --allow-no-sex
代码解释:
- –file为plink的文件名的前缀
- –pheno 为plink分析GWAS的表型数据,注意前两列为FID IID,第三列为分析的性状
- –assoc 利用的是LM模型
- –out 输出文件名
- –allow-no-sex 因为数据中没有性别信息,需要加上这个参数,允许没有性别的数据
运行日志:
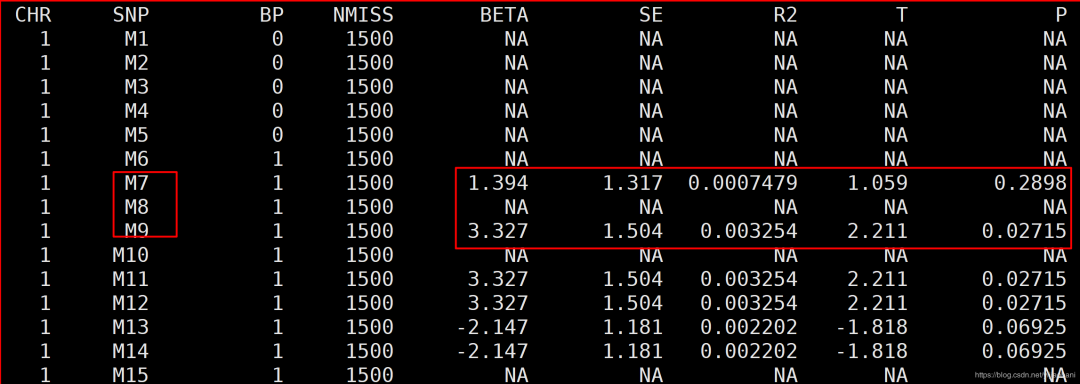
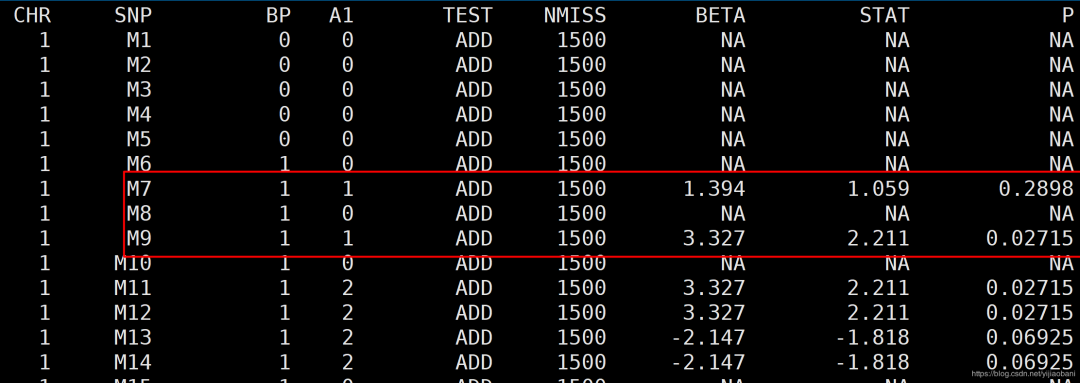
结果文件:re.qassoc
注意,这里的SNP没有质控,有很多SNP没有分型,所以结果有很多NA,不过这里可以看出assoc分析GWAS的结果,其中SNP中M7和M9的结果:
- M7,beta值为1.394,se为1.317,R2为0.000749,T值为1.059,P值为0.2898
- M9,beta值为3.327,se为1.504,R2为0.003254,T值为2.211,P值为0.02715
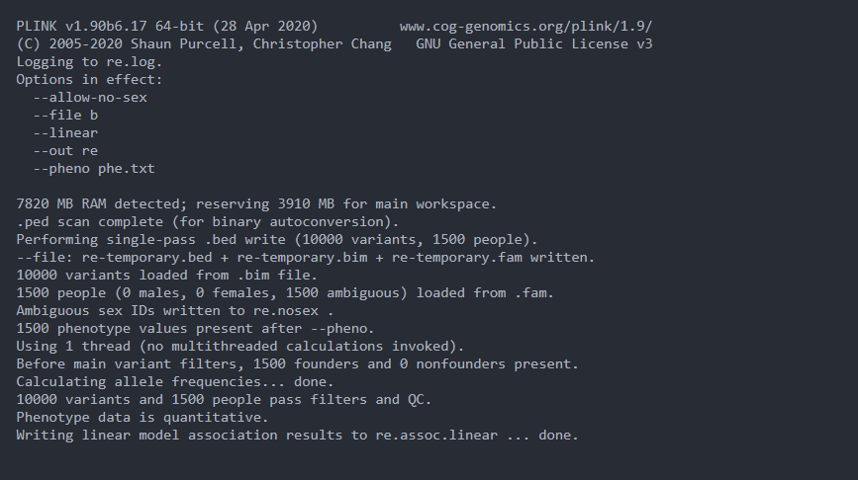
4.plink利用linear进行LM的GWAS分析
这里,利用linear和上面的assoc命令类似,将linear代替assoc即可。一般来说,linear可以支持协变量,assoc不支持协变量。assoc运行速度快于linear。
plink --file b --pheno phe.txt --allow-no-sex --linear --out re
运行日志:
结果文件:re.assoc.linear
结果和assoc的结果完全一致!
5.使用R语言的一般线性回归比较结果
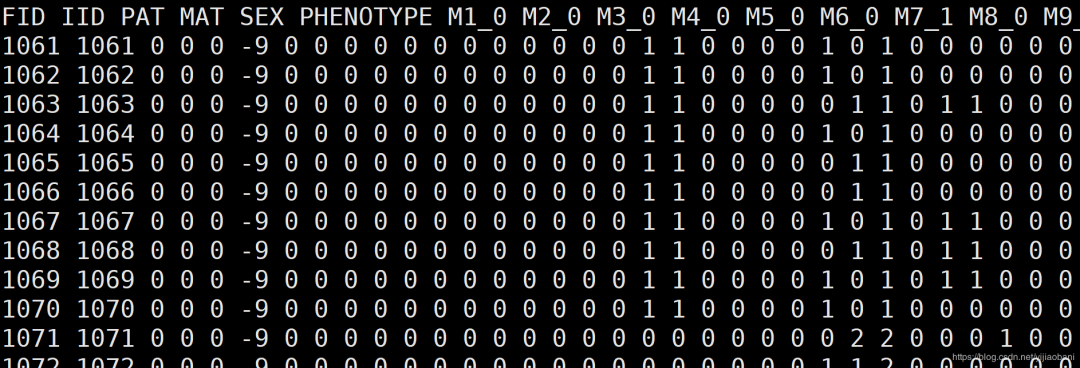
这里,需要将plink转化为012的形式,可以使用recodeA参数:
plink --file b --recodeA --out c
结果文件:c.raw
编写R程序:
library(data.table)
geno = fread("c.raw",header=T)
geno[1:10,1:20]
phe = fread("../phe.txt")
head(phe)
dd = data.frame(phe$V3,geno[,7:17])
head(dd)
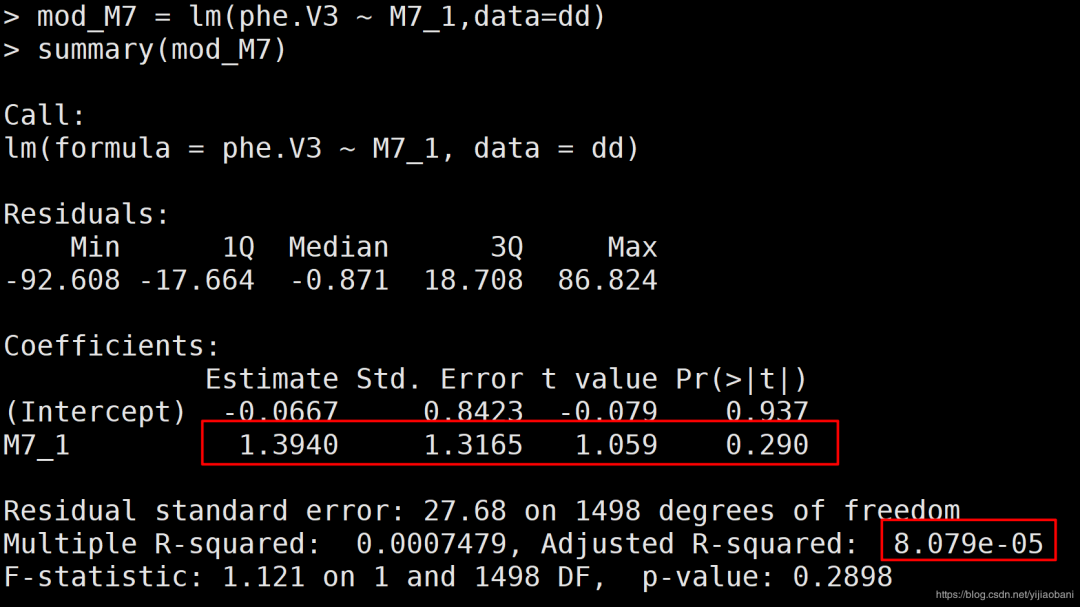
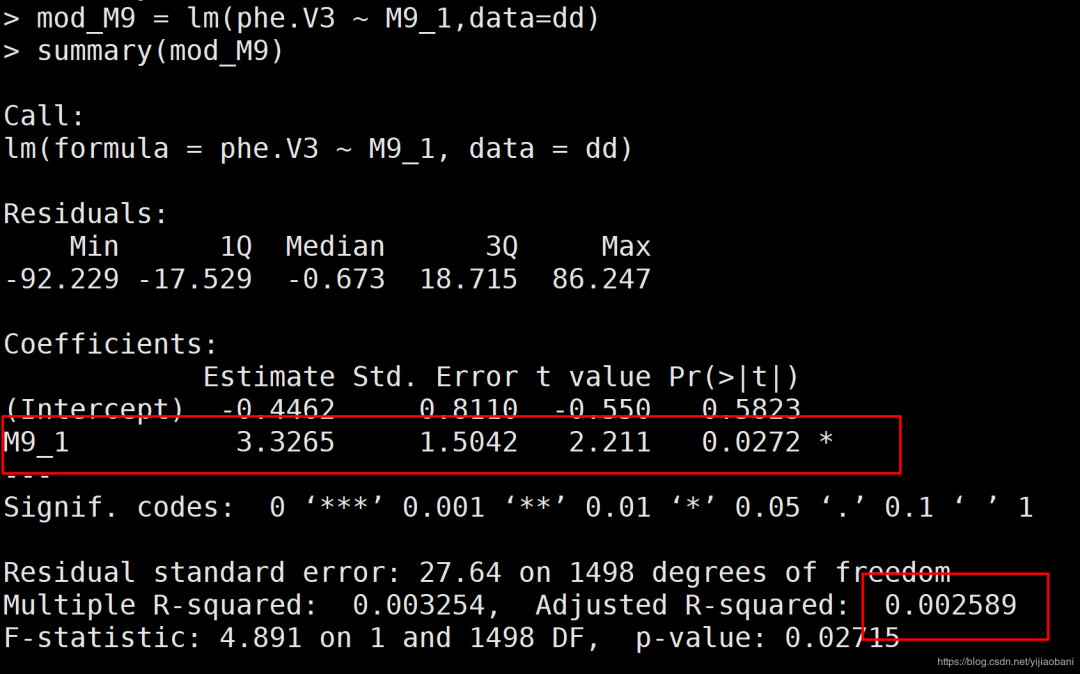
mod_M7 = lm(phe.V3 ~ M7_1,data=dd)
summary(mod_M7)
可以看出R语言lm的结果中:M7: beta为1.3940, se为1.3165,t值为1.059,p值为0.290; M9:beta为3.3265,se为1.5042,t值为2.211,p值为0.0272。
对比assoc的结果:
可以看出,两者结果完全一致。
6.Note
1.GWAS分析中,plink进行LM分析,SNP转化为012, 0是major,2是minor,这顺序是不能变的,你如果要手动进行回归分析,需要将SNP的分型进行转化,而不是粗暴的将其替换为0,1,2.
2.R在进行单个SNP的分析时,0,1,2是作为数值进行的回归分析,而不是因子。