资料来源:公众号:育种数据分析之放飞自我
本篇主要是介绍一般线性模型(LM)中的linear参数,考虑数值协变量,然后将结果与R语言编程结果比较。
1.协变量文件整理
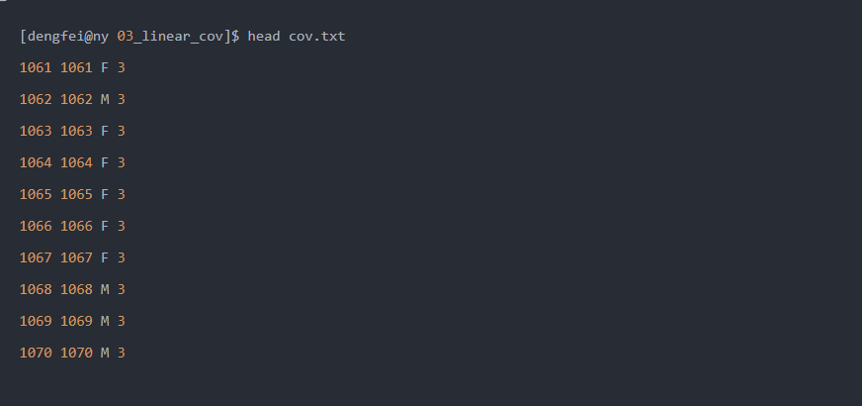
第一列为FID ,第二列为ID ,第三列以后为协变量(注意,只能是数值,不能是字符!)
这里协变量文件为:
这里第三列为性别,第四列为世代,为了方便操作,我们将世代作为数值,直接进行协变量分析.
2.数值协变量
awk '{print $1,$2,$4}' cov.txt >cov1.txt
数据如下:
3.进行数值协变量GWAS分析LM模型
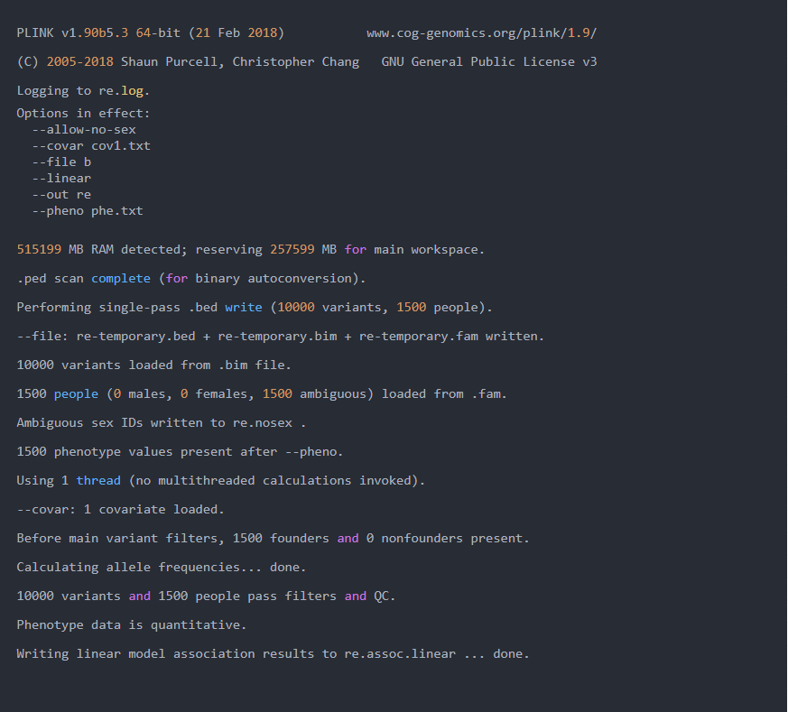
代码:
plink --file b --pheno phe.txt --allow-no-sex --linear --covar cov1.txt --out re
日志:
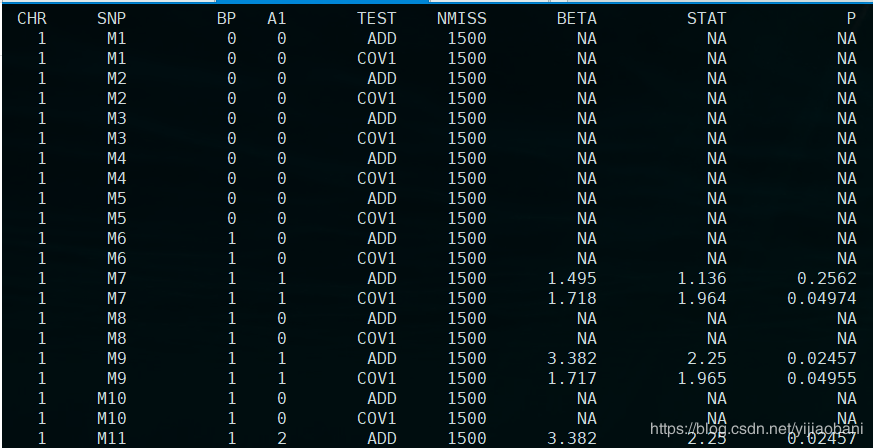
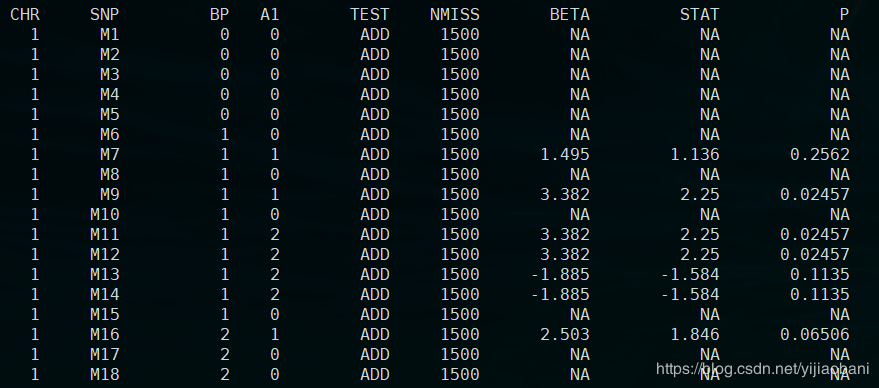
结果文件:re.assoc.linear
这里的结果包括协变量的检验,我们不需要输出协变量结果,可以加上参数:–hide-covar
5.使用R语言进行结果比较
library(data.table)
geno = fread("c.raw")
geno[1:10,1:10]
phe = fread("phe.txt")
cov = fread("cov.txt")
dd = data.frame(phe$V3,cov$V4,geno[,7:20])
head(dd)
str(dd)
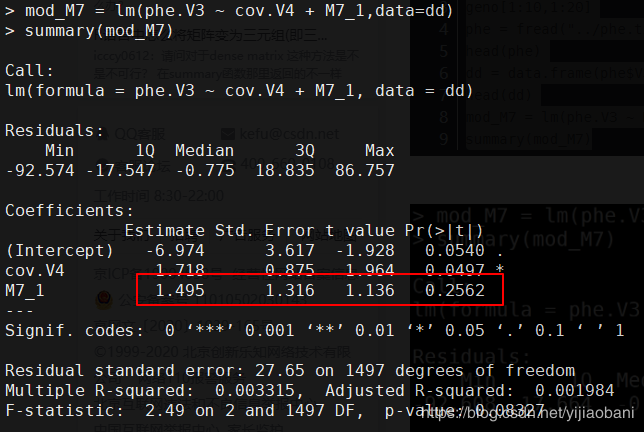
mod_M7 = lm(phe.V3 ~ cov.V4 + M7_1,data=dd)
summary(mod_M7)
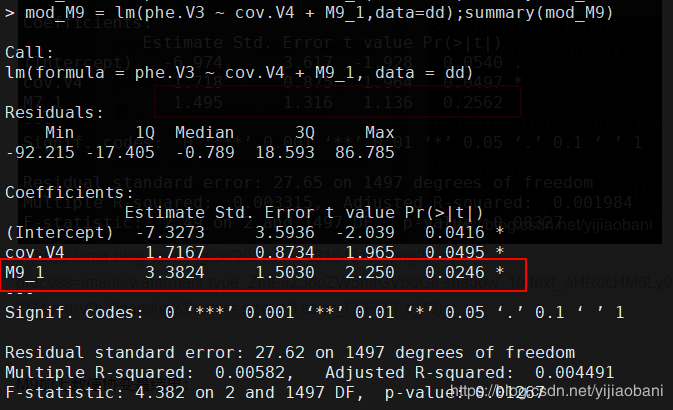
mod_M9 = lm(phe.V3 ~ cov.V4 + M9_1,data=dd);summary(mod_M9)
M7加上数值协变量结果:
M9加上数值协变量结果:
结果完全一致。